Evoluzione dei metodi di sequenziamento del DNA
Riduzione dei tempi e dei costi di sequenziamento
Durante gli ultimi anni, i metodi di sequenziamento del DNA sono andati incontro a una evoluzione senza precedenti. La produzione dei dati è cresciuta in maniera esponenziale. Il costo per nucleotide si trova in continua caduta spostando l’ago della bilancia della difficoltà verso l’analisi bioinformatica.
Negli ultimi anni i sequenziatori di DNA sono andati incontro a una evoluzione tecnologica vertiginosa. La prima generazione di sequenziatori capillari, basati principalmente sul metodo di Sanger, ottenevano una sequenza per reazione. Gli attuali metodi di sequenziamento parallelo di seconda (Illumina, IonTorrent) e terza generazione (Oxford Nanopore Technologies, Pacific Bioiscience), anche noti come Next Generation Sequencing (NGS), producono milioni di sequenze in una unica reazione. I metodi NGS permettono inoltre processare più campioni simultaneamente (multiplexing).
Il costo dell’acquisizione di queste nuove macchine è relativamente elevato e non è più giustificabile per il singolo laboratorio di piccole o medie dimensione. Questi nuovi sequenziatori diventano generalmente una prerogativa di grandi laboratori o servizi centralizzati. È anche importante considerare che i metodi NGS hanno ridotto di molto i tempi di produzione dei dati. Progetti di sequenziamento di genomi, basati sul metodo di Sanger, potevano durare dei mesi o anni. Con l’arrivo dei metodi NGS, gli stessi progetti si risolvono oggi in pochi giorni (o addirittura ore). Tutto ciò ha portato a una “democratizzazione” del sequenziamento del DNA, rendendolo finalmente accessibili a tutti i laboratori.
Se questa prima parte della produzione dei dati non rappresenta più un ostacolo, il campo di battaglia si sposta sull’analisi bioinformatica dei dati provenienti dal sequenziamento del DNA.
Sistema operativo Linux e programmazione per l’analisi bioinformatica
Usando il terminale e le linee di comando
Quando le sequenze arrivano finalmente ai computer di un ricercatore, questi si troverà di fronte a files giganteschi, fatti di nucleotidi e strani simboli da copiare, pulire e riordinare solo per iniziare a lavorarci sopra. Tutto questo in un sistema operativo Linux, considerato uno standard per l’analisi bioinformatica.
Il ricercatore si trova di colpo a dover lavorare con programmi che funzionano esclusivamente mediante linee di comando. Deve imparare a destreggiarsi in un ambiente Linux e ad inserire correttamente i comandi in un terminale (Figura 1). I problemi dei biologi (e simili) che, in fin dei conti finora si risolvevano sui libri dell’università, diventano oggi quasi esoterici. Il biologo deve iniziare a conoscere i flussi di lavoro per eseguire un controllo di qualità delle sequenze per poi, finalmente, portare avanti processi di:
- assemblaggio di genomi de-novo,
- mapping (Figura 2),
- ricerca di mutazioni,
- annotazione di genomi,
- analisi funzionali e tassonomiche di transcrittomi e metagenomi,
- analisi tassonomiche di popolazioni microbiche complesse,
- eccetera.
Le possibilità di analisi offerte dalla bioinformatica diventano praticamente infinite.
Il passaggio a un sistema basato sul terminale è una tappa obbligata. I dati su cui si lavora non si possono leggere con i normali programmi di offimatica a cui si è abituati. I files che procedono dal sequenziamento del DNA sono incredibilmente grandi e la memoria RAM (Random Access Memory) dei computer comincia a giocare un ruolo importante nel leggere o riassumere il prodotto del sequenziamento. Ci si ritrova inoltre a dover spesso eseguire lavori lunghi e ripetitivi per cui si devono pianificare automazioni “in loop“. Si devono creare dei cicli che, con poche linee di comando, possono eseguire analisi ripetitive su centinaia di campioni (Perkel, 2021).
Imparare almeno un linguaggio di programmazione
Quando ci si addentra nell’analisi bioinformatica di dati NGS, ci si accorge di come il fulcro della ricerca si sia spostato dai banchi di laboratorio ai computer. Un bioinformatico si accorge di poter lavorare via Internet da qualsiasi postazione fisica. Può abbordare temi di diversa natura, usando i dati prodotto in laboratorio, o attingendo alla onniscenza collettiva offerta dai grandi database di pubblico accesso. Si pensi che alla fine di maggio 2021 è stata pubblicata l’ultima versione di GenBank (release 243.0) con 14.03 trilioni di nucleotidi e 2.40 bilioni di records!
Una delle meraviglie che ci offre la bioinformatica è l’apertura a un mondo di possibilità di cui non si conosceva l’esistenza. Questo mondo, ci si svela non appena cominciano a dominare alcuni linguaggi di programmazione come Perl, Python o R. Imparare almeno uno di questi linguaggi è un passo fondamentale per tutti i nuovi bioinformatici. Il bioinformatico ne farà un uso continuo insieme a un arsenale di risorse per lavorare dai dati grezzi fino alla risoluzione dell’ipotesi di partenza. La bioinformatica si alimenta dei dati prodotti in laboratorio, o di quelli già pubblicati, e li digerisce mediante diversi canali di analisi.
Si inizia generalmente da una analisi primaria che traduce i dati dal sequenziamento in dati genomici (un assemblaggio o un mapping) o in tabelle riassuntive che descrivono distribuzioni di annotazioni funzionali, conteggi di unità tassonomiche, eccetera. Questa parte si avvale di flussi di lavoro standardizzati e accettati a livello internazionale che appartengono, come lo stesso Linux, al mondo Open Source. È anche importante considerare che, con l’aumento della complessità dei dati prodotti, sono nati dei formati standard e dei consorzi che stabiliscono le linee guida per la descrizione di campioni, dei metodi e dei processi analitici (Genomic Standards Consortium, Yilmaz P. et al., 2011).
La bioinformatica per arrivare alla conclusione
Una volta ottenuti i dati primari, si procede dunque alla parte più analitica. Le nozioni di genomica e di statistica, proprie del bagaglio formativo di un biologo (e simili), permetteranno di risolvere l’ipotesi di partenza e arrivare alla conclusione. In questa seconda fase, il bioinformatico inizia a destreggiarsi fra linguaggi matematici (generalmente R), o programmi di visualizzazione per genomi come Artemis o IGV (Figura 2). Può anche trovare nuove vie per arrivare al risultato finale. I nuovi metodi e i programmi sviluppati possono diventare oggetto di ulteriori pubblicazioni.
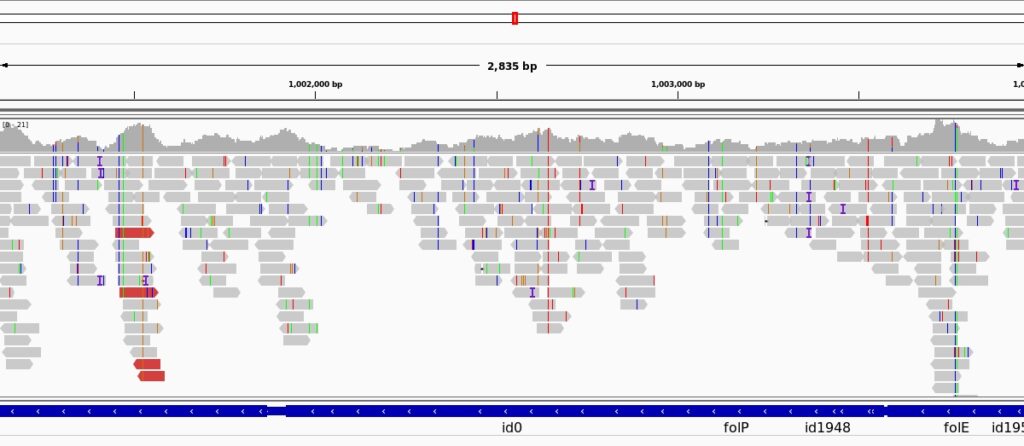
Indipendentemente dall’origine dei dati, i bioinformatici si trovano inevitabilmente alla fine della catena di montaggio. Se l’avvento dei servizi centralizzati hanno semplificato la produzione massiva dei dati, l’analisi bioinformatica diventa il nuovo collo di bottiglia. Ci troviamo in una situazione in cui pochi “guru” della biologia con nozioni di informatica (o al contrario) lavorano su un fiume in piena di dati genomici. Questo fa di un bioinformatico un bene prezioso in qualunque laboratorio a cui non smetteranno di arrivare nuovi file da analizzare e nuove sfide da superare.
Consigli utili per principianti (microbiologi, biotecnologi, x-ologi)
Perle di saggezza per i biologi che vogliono iniziarsi alla bioinformatica per NGS:
- Iniziare a lavorare con un sistema operativo basato su Unix (Linux/Mac).
- Leggere e studiare gli errori (o copiarli e incollarli su qualunque motore di ricerca).
- Imparare BASH come linguaggio per parlare al computer.
- Imparare un linguaggio utile per il calcolo e la rappresentazione dei dati (R, Python, Perl).
Sull’autore: Dr. Giuseppe D’Auria, PhD. (ORCID, gidauria), responsabile del Servizio di Bioinformatica della fondazione FISABIO, Valencia, Spagna. Progetto “Microonstream: Life under the microscope” (YT, Twitter).
Fonti
Pubblicazioni:
- Perkel JM. Five reasons why researchers should learn to love the command line. Nature. 2021 Feb;590(7844):173-174.
- Yilmaz P, Kottmann R, Field D, et al. Minimum information about a marker gene sequence (MIMARKS) and minimum information about any (x) sequence (MIxS) specifications. Nat Biotechnol. 2011 May;29(5):415-20.
Linguaggi di programmazione e programmi citati:
- BASH: https://tiswww.case.edu/php/chet/bash/bashtop.html
- Perl: https://www.perl.org/
- Python: https://www.python.org/
- The R Project for Statistical Computing: https://www.r-project.org/
- GenBank release 243.0: https://ncbiinsights.ncbi.nlm.nih.gov/2021/06/03/genbank-release-243/
- Lubuntu, Distribuzione Linux basata su Ubuntu: https://lubuntu.net/
- Artemis genome browser and annotation tool: https://www.sanger.ac.uk/tool/artemis/
- Integrative Genomics Viewer: https://software.broadinstitute.org/software/igv/