Scoperta e caratterizzazione
Marzo 2020: viene caratterizzata per la prima volta, tramite criomicroscopia elettronica, la proteina spike del novel coronavirus (2019 n-CoV), ovvero proprio quel virus che aveva catturato l’attenzione di tutto il mondo scientifico e non. Ripercorriamo insieme gli eventi più importanti:
Dicembre 2019: le autorità cinesi riportano numerosi casi di “polmoniti anomale” in Cina, soprattutto a Wuhan.
Gennaio 2020: il governo cinese accerta che i casi anomali erano causati da un virus appartenente alla stessa famiglia di SARS-CoV e MERS.
Marzo 2020: l’OMS dichiara la pandemia da SARS-CoV-2.
Nello stesso mese e nel seguente, due gruppi di ricerca hanno determinato la struttura della proteina spike mediante criomicroscopia elettronica, giungendo così al medesimo risultato ma in maniera indipendente. In particolare, la proteina spike è caratterizzata da due stati conformazionali dai quali dipende l’accesso del virus nella cellula rendendola un target essenziale per la neutralizzazione mediata da anticorpi. Inoltre la sua caratterizzazione a livello molecolare ha fornito numerose informazioni per la produzione di vaccini a base di acidi nucleici.
Struttura molecolare
La glicoproteina S “Spike” del SARS-CoV-2 è una proteina transmembrana responsabile del caratteristico aspetto “a corona” dei coronavirus, dalla quale questi ultimi prendono il nome. Approfondiamo insieme le caratteristiche strutturali e molecolari di questa proteina:
Con un peso di 180-200 kDa, la proteina spike è composta da 1273 amminoacidi che contribuiscono a formare diversi domini strutturali e funzionali.
In particolare, la “Spike” è composta da un peptide segnale (aminoacidi 1-13 N-terminale), e due subunità aminoacidiche S1 ed S2.
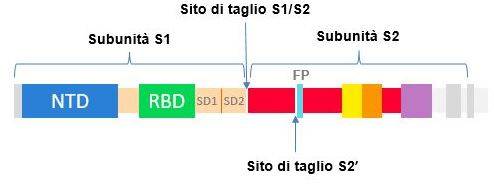
- La subunità S1 (residui 14-685) contiene: una sequenza di aminoacidi nota come RBD, receptor binding domain, fondamentale per l’interazione del virus con il suo recettore presente sulla membrana della cellula ospite ed un dominio extracellulare N-terminale.
- La subunità S2 (residui 686-1273) comprende i residui amminoacidici responsabili della fusione finale del virus nella cellula ospite, ovvero: il peptide di fusione (FP), HR1, HR2, il dominio citoplasmatico e quello transmembrana (TM) che àncora la proteina alla membrana del virus; ed infine, un piccolo dominio C-terminale.
È necessario tenere in considerazione che da un punto di vista molecolare, la proteina “Spike” di SARS-CoV-2 è costituita da tre “porzioni” uguali tra loro dette protomeri, le quali interagendo tra loro formano la struttura finale della “Spike”, ovvero una proteina trimerica transmembrana. Ne consegue che ogni protomero è formato dai domini strutturali e funzionali appena descritti.
Strutturalmente, la glicoproteina S “Spike” può assumere diverse conformazioni a seconda dell’interazione e della posizione assunta dal dominio di legame con il recettore (RBD) situato nella subunità S1:

Per permettere l’ingresso del virus nella cellula, il dominio RBD della subunità S1 deve andare incontro ad una modificazione conformazionale che potremmo definire “a cerniera”: questi movimenti rendono visibili i siti di interazione del dominio RBD con il suo recettore, determinando l’inizio dell’internalizzazione del virus nella cellula ospite.
Quindi, ogni protomero della proteina Spike può esistere in due conformazioni a seconda della posizione assunta dal dominio di legame del recettore (RBD), avremo perciò:
- La conformazione “su” o attiva, quando RBD è pronto ad interagire con il suo recettore.
- La conformazione “giù” o nativa, quando RBD nasconde i siti di interazione con il recettore.
Meccanismo d’azione
Analizzata la struttura molecolare della glicoproteina S “Spike”, possiamo descrivere a questo punto il suo ruolo cruciale nell’infezione da SARS-CoV-2.
In particolare, la proteina “Spike” è essenziale per le prime due fasi del ciclo di replicazione ovvero l’ingresso e la fusione del virus all’interno della cellula ospite. Approfondiamo di seguito questi primi step:
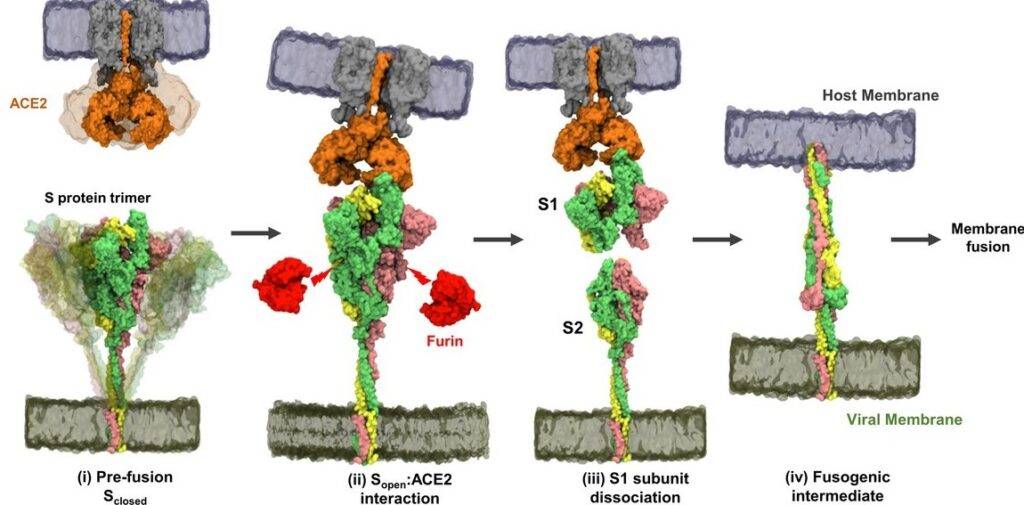
Prima dell’infezione della cellula, la CoV-2-S si trova in forma di precursore inattivo ovvero la proteina è nella sua conformazione nativa dove il dominio di legame con il recettore nasconde i suoi siti di interazione con il recettore specifico.
Durante l’infezione virale, le proteasi cellulari attivano la proteina “Spike” nella sua conformazione attiva effettuando un taglio tra le subunità S1 ed S2 visibile nella Fig.1. Infatti, al confine tra queste due subunità, vi è una sequenza di quattro aminoacidi che consente il legame di una specifica proteasi umana chiamata furina, come possiamo vedere nella Fig.3, responsabile del clivaggio delle subunità S1 ed S2.
A questo punto entrambe le subunità possono mediare il rispettivo e specifico ruolo:
- La subunità S1, attraverso il dominio RBD (receptor binding domain) nella conformazione “su”, lega il recettore ACE2 localizzato sulla membrana plasmatica della cellula ospite, il quale rappresenta la porta d’ingresso per il virus nella cellula. L’interazione tra il dominio RBD e il recettore ACE2 induce dei cambiamenti conformazionali nella proteina spike, fondamentali per avvicinare la membrana plasmatica e il virione al fine di rendere possibile la fusione del virus.
- La subunità S2 è la responsabile del processo di fusione finale, attraverso le sue specifiche sequenze amminoacidiche.
Funzioni biologiche
Data la struttura molecolare ed il meccanismo d’azione della glicoproteina S “Spike”, vediamo insieme le sue funzioni biologiche.
La prima funzione svolta dalla CoV-2-S è sicuramente quella di struttura. In particolare, la “Spike” è una delle proteine codificate dai geni strutturali del SARS-CoV-2 e responsabili della formazione del virione, il quale è costituito inoltre dalla la proteina N (del nucleocapside), la proteina E (dell’envelope), la proteina M (di membrana).
La “Spike” riveste un ruolo di fondamentale importanza nell’infezione da SARS-CoV-2 in quanto chiave d’ingresso del virus, senza la quale sarebbe impossibile mediare il processo di infezione e replicazione nelle cellule eucariotiche.
Applicazioni biotecnologiche, mediche ed industriali
La proteina “Spike” ha rappresentato il primo target nello sviluppo dei vaccini attualmente in commercio. In particolare, proprio la descrizione della sua struttura molecolare mediante criomicroscopia elettronica, ha permesso di comprendere al meglio non solo la composizione atomica della proteina ma anche e soprattutto le sue diverse conformazioni, indirizzando in questo modo il mondo scientifico nella sintesi e sviluppo di nuovi vaccini.

La risposta immunitaria elicitata dai vaccini attualmente in commercio, consente infatti di neutralizzare il virus impedendo il legame del dominio RBD della glicoproteina con il suo recettore ACE2, prevenendo così l’infezione.
L’evoluzione della proteina Spike
Nel corso della pandemia, la veloce diffusione del SARS-CoV-2 ha portato all’insorgenza, in alcuni paesi, di diverse varianti che gradualmente si sono a loro volta diffuse nel resto del mondo. Le diverse varianti sono caratterizzate, dal punto di vista epidemiologico, da una differente trasmissibilità e patogenicità ma soprattutto da alcune variazioni amminoacidiche che si ripercuotono sulla funzione svolta dal dominio che ha acquisito la mutazione.
Gli scienziati hanno osservato le diverse mutazioni distribuite su tutta la catena aminoacidica. In particolare, le più comuni sono localizzate proprio a livello della subunità S1 della glicoproteina S “Spike”:
- In posizione 614, la mutazione sembra controllare la stabilità della porzione superiore del trimero di “Spike”;
- In posizione 681, la mutazione coinvolge il segmento flessibile tagliato dalla furina (vedi Fig.3) durante il processo di infezione virale, di conseguenza sembra correlare con una maggiore infettività.
Tutte le varianti affermate durante il corso della pandemia includono queste due mutazioni nella catena aminoacidica. Tuttavia presentano anche molte altre mutazioni localizzate a livello delle altre proteine virali sopracitate, conferendo nuove o migliorate funzioni biologiche al virus.
Fonti
- Huang Y, Yang C, Xu XF, Xu W, Liu SW. Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacol Sin. 2020 Sep;41(9):1141-1149
- https://sibbm.zanichelli.it/italiano/2020/08/23/infezione-biologia-strutturale/
- Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS. Cryo-EM Structure of the 2019-nCoV Spike in the Prefusion Conformation. bioRxiv [Preprint]. 2020 Feb 15:2020.02.11.944462. doi: 10.1101/2020.02.11.944462. Update in: Science. 2020 Mar 13;367(6483):1260-1263. PMID: 32511295; PMCID: PMC7217118.
- https://www.pianetachimica.it/mol_mese/mol_mese_2021/12_Varianti_della_proteina_Spike/Varianti_proteina_Spike.htm
Crediti immagine in evidenza:
2 commenti su “Glicoproteina S “Spike” (SARS-CoV-2)proteina”