CARATTERISTICHE
L’Hepatitis C Virus, conosciuto più comunemente come HCV, è un virus a RNA a singolo filamento appartenente alla famiglia dei Flaviviridae. Tale virus è conosciuto per essere l’agente eziologico dell’epatite C, patologia che comporta una infiammazione del fegato con conseguente forma cronica di danno epatico.
A livello mondiale, ad oggi sono circa 150 milioni le persone affette dal virus HCV, con un’ampia variabilità della distribuzione geografica. Molto spesso però i dati epidemiologici potrebbero non essere del tutto congruenti alla reale diffusione del virus, poiché la sua latenza non permette una diagnosi precoce ma solo quando sono presenti specifiche manifestazioni cliniche. La via di trasmissione del virus è parenterale, attraverso il sangue e gli emoderivati: trasfusioni, utilizzo di aghi o strumentazioni chirurgiche non sterilizzate, scambio promiscuo di siringhe, sono tutte modalità che permettono il diffondersi dell’infezione con interesse epatico; l’epatite C, infatti, specialmente negli anni ’80 – ’90, in cui non erano presenti i test sierologici per i donatori di sangue, rappresentava la causa maggiore di epatite post – trasfusionale, venendo chiamata “epatite non A e non B“.
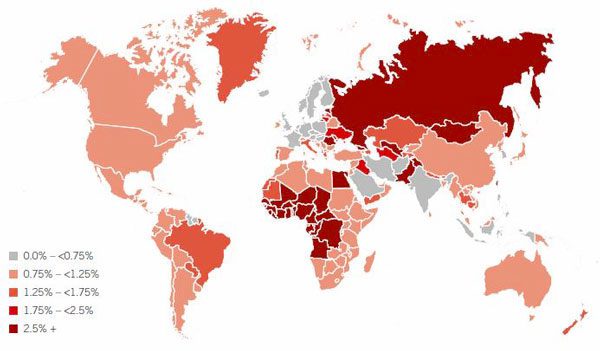
Figura 1 – Distribuzione geografica del virus
FILOGENESI
| Dominio | Acytota |
| Gruppo | Gruppo IV (ssRNA+) |
| Famiglia | Flaviviridae |
| Genere | Hepacivirus |
| Specie | Hepatitis C Virus |
MORFOLOGIA STRUTTURALE
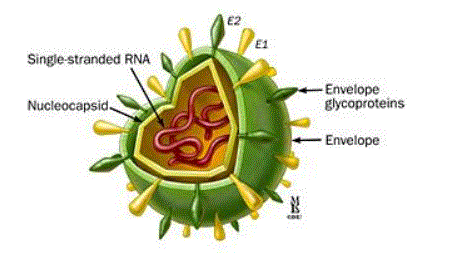
Il virione del virus HCV, ossia la singola particella virale, presenta un diametro di circa 55-65 nm ed è provvisto di un genoma a RNA a singolo filamento a polarità positiva di 9.5 kb alle cui estremità sono presenti due regioni non codificanti a funzione regolatrice (5′ UTR e 3′ UTR) che fiancheggiano un’ampia regione centrale codificante una poliproteina virale processata da proteasi cellulari e virali, dando origine alle proteine che compongono il genoma di HCV: tre proteine strutturali quali il Core (C) e le glicoproteine dell’envelope virale, E1 ed E2 e proteine non strutturali quali NS2, NS3, NS4A, NS4B, NS5A, NS5B che intervengono nella replicazione virale.
L’RNA del virus è racchiuso in un capside icosaedrico (core) al cui esterno è presente il doppio strato fosfolipidico contentente E1 ed E2. Di notevole rilevanza, oltre alle proteine appena descritte, è la piccola proteina P7. Quest’ultima è un piccolo polipeptide di membrana con attività di canale ionico, ancora oggi oggetto di studio, che presenta un ruolo importante nell’assemblaggio e infezione del virus alle cellule specifiche.

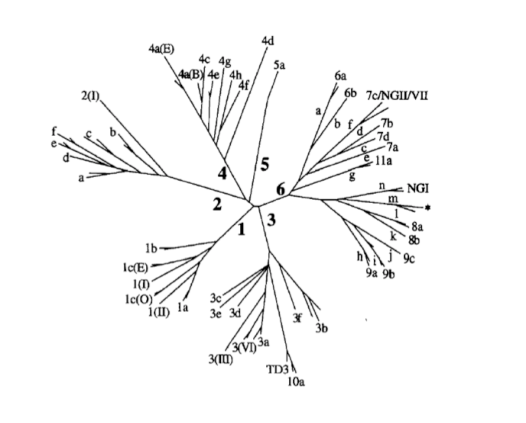
Il virus entra nelle cellule bersaglio, gli epatociti, mediante endocitosi mediata da recettore: una volta endocitato rilascia il proprio RNA a polarità positiva che, funzionando già come mRNA, si lega ai ribosomi iniziando la traduzione, con successiva produzione delle proteine virali necessarie alla replicazione: il genoma viene replicato a partire dalla proteina NS5B, una RNA-polimerasi RNA-dipendente. Nella sua attività replicativa si lega principalmente ai recettori CD81, espressi sia sugli epatociti e sui linfociti B: può inoltre legarsi alle LDL ed utilizzarle, assieme alle VLDL, come rivestimento eludendo l’attacco del sistema immunitario ed iniziando così la sua attività infettiva nell’organo colpito. Al termine del ciclo replicativo il virione viene rilasciato per esocitosi. Inoltre, una peculiare caratteristica del virus HCV è la sua ampia ricombinazione genetica, che gli conferisce la possibilità di avere diversi genotipi che in clinica vengono suddivisi in 1, 2, 3, 4, 5, 6: tali genotipi differenziano fra loro per distribuzione geografica ma soprattutto per sottoclasse e maggiore virulenza: infatti, tra questi, sono conosciute le principali sottoclassi 1a, 1b, 2a, 2b, 3a, 4c e 4d. Il più diffuso, nonché il più virulento è l’1b.

PATOGENESI
Il danno del virus HCV è principalmente immuno-mediato: esso, infatti, prima di giungere al suo sito d’elezione, ossia il fegato, infetta i linfociti T e B. La velocità di replicazione del virus è molto alta (1×1012 virioni al giorno circa). La risposta immunitaria innata si manifesta inizialmente con la produzione di citochine ma, alcune proteine codificate da HCV come il core, NS3A, NS4A, NS4B permettono al virus di eludere le difese immunitarie innate ed iniziare così una attiva replicazione virale: ciò comporta, nella maggioranza dei casi, la persistenza e la cronicizzazione dell’infezione, con lesioni citopatiche ben evidenti. Tale fenomeno trova riscontro in due principali manifestazioni cliniche:
- cirrosi epatica, con perdita del tessuto epatico sano e successiva sostituzione con tessuto fibrotico, che comporta una perdita progressiva delle molteplici funzioni epatiche con comparsa di ittero, ipertensione portale, ascite, varici esofagee, encefalopatia epatica e nei casi più gravi, se non è presente un intervento imminente, coma epatico;
- epatocarcinoma, il più frequente tumore di origine epatica conseguente alla cirrosi, prima causa di trapianto di fegato.
METODI DI IDENTIFICAZIONE
L’identificazione e successiva diagnosi del virus si basa sulla ricerca quantitativa dell’antigene HCV (HCVAg) mediante immunodosaggio in chemiluminescenza e degli anticorpi specifici di tipo IgG contro il virus (HCVAb) nel siero. Per discriminare la presenza di anticorpi si utilizza un immunodosaggio di terza generazione che prevede l’utilizzo di regioni antigeniche del virus quali il core, NS3, NS4A, NS4B e NS5A.
Oltre alle indagini sierologiche, di estrema importanza nonché di conferma in caso di positività a queste ultime, si procede alla biologia molecolare in infettivologia molecolare con lo studio dei marcatori diretti di infezione attraverso la ricerca dell’HCV RNA mediante prima estrazione automatizzata del genoma virale e successiva RT (reverse transcriptase-polymerase chain reaction), poiché il genoma del virus è a RNA e per essere amplificato in PCR necessita della trascrizione inversa il cui prodotto finale sarà il cDNA. Tale prodotto verrà poi amplificato in Real Time PCR per avere la quantizzazione finale delle copie del genoma di HCV.
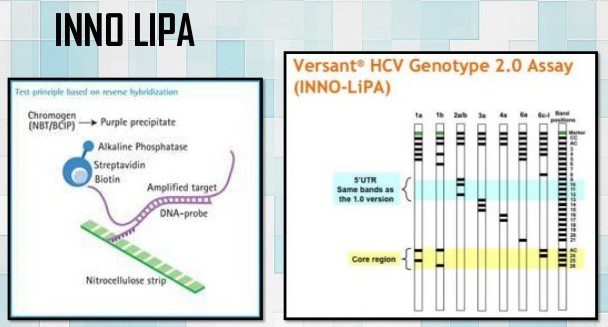
Di estrema importanza, oltre alla ricerca molecolare del virus, è la successiva genotipizzazione, di particolare rilevanza per la messa in atto di un percorso terapeutico mirato. La genotipizzazione è svolta con una metodica di ibridazione inversa del DNA amplificato a sonde oligonucleotidiche specifiche adese su delle strisce di nitrocellulosa. La comparsa, dopo incubazione, di determinate colorazioni su tali strisce, permetterà la discriminazione di un certo genotipo rispetto ad un altro.
TERAPIA
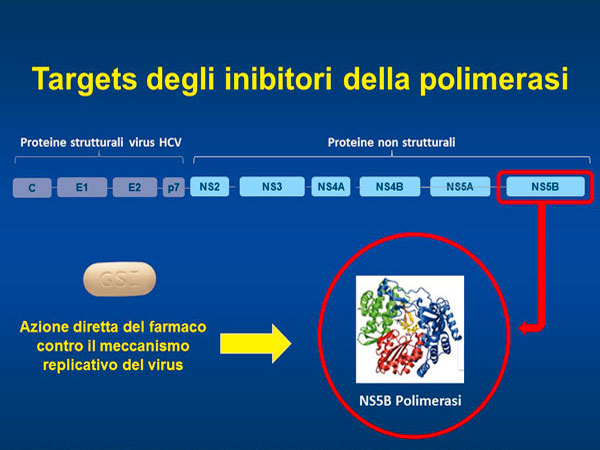
Fino al 2013, lo schema terapeutico di elezione per il trattamento e l’eradicazione del virus HCV era l’utilizzo combinato dell’interferone e della ribavirina. Dal 2013, è iniziato l’utilizzo di nuovi farmaci ad attività antivirale diretta e specifica denominati Directly Acting Antivirals – DAA il cui meccanismo di azione è basato principalmente sull’inibizione dell’attività replicativa del virus, agendo direttamente sulle proteine che intervengono in quest’ultimo processo, come NS3, NS5A ed NS5B. Tali farmaci, quali ad esempio il Sofosbuvir, Simeprevir ed il Daclatasvir (utilizzati sempre in duplice terapia) sono risultati molto più efficaci e tollerati dai pazienti in cura, poiché andando ad agire in maniera diretta e specifica sui complessi di replicazione del virus, garantiscono una capacità di guarigione di oltre il 90 % anche nei pazienti affetti dal genotipo 1b, da sempre il più difficile da eradicare. Tali farmaci sono in continua sperimentazione, il cui fine ultimo – non essendo presente un vaccino che possa eliminare a monte la possibilità di contagio del virus – è quello di rendere l’epatite C una patologia definitivamente curabile e debellare così l’Hepatitis C Virus.
Priscilla Caputi
Fonti
- https://microbenotes.com/hepatitis-c-virus/
- https://www.epac.it/
- http://old.iss.it/pato/?lang=1&id=58&tipo=15
- Kenneth J. Ryan, C. George Ray. Sherris, Microbiologia Medica. The McGraw Hill Companies, 2017. Edizioni Mediche Scientifiche Internazionali.
- Atoom, A. M., Taylor, N. G., & Russell, R. S. (2014). The elusive function of the hepatitis C virus p7 protein. Virology, 462, 377-387.
- Saint, N., Montserret, R., Chipot, C., & Penin, F. (2009). Structural and functional analysis of the HCV p7 protein. In Hepatitis C (pp. 125-143). Humana Press.