“Ciao Gianluca, grazie per concedere questa intervista per microbiologiaitalia.it. Parlaci un po’ di te per chi tra i lettori non ti conoscesse”
Ciao Francesco, grazie a voi di microbiologiaitalia.it per questa opportunità!
Io mi chiamo Gianluca Tomasello, e sono laureato in Biotecnologie. È durante il tirocinio triennale che ho iniziato ad utilizzare i primi software per la visualizzazione molecolare in 3D. Dopo la laurea triennale, non ero convinto di voler continuare con il percorso di studi. Quindi ho deciso di prendermi 6 mesi di tempo per decidere il da farsi. Durante questo periodo ho svolto un tirocinio extracurriculare nello stesso laboratorio dove ho lavorato in triennale per continuare a studiare e informarmi nell’ambito della visualizzazione molecolare.
Dopo di che ho deciso di continuare con la magistrale dove ho potuto ampliare le mie conoscenze in ambito fisico-chimico e computazionale. Durante il tirocinio, ho anche avuto l’opportunità di trascorrere un periodo all’Istituto italiano di tecnologia di Genova per apprendere tecniche computazionali più avanzate come la dinamica molecolare.
Questo periodo mi ha motivato ancora di più a continuare il percorso intrapreso e così ho deciso di continuare a studiare e informarmi nel settore della visualizzazione molecolare anche dopo la laurea.

“Sei un illustratore molecolare, probabilmente molti non conoscono questo lavoro e quali competenze servono. Puoi parlarcene?”
Si, mi definisco un illustratore molecolare, mi piace essere specifico perché mi sono grandemente specializzato nel settore “molecolare” appunto. Il disegno a mano libera non fa parte delle mie competenze, io lavoro interamente a computer utilizzando vari software (scientifici e non) per illustrare modelli tridimensionali di molecole e macromolecole biologiche come proteine e DNA.
Per poter essere un illustratore molecolare si devono acquisire varie competenze in ambiti differenti. Per prima cosa servono ampie conoscenze in ambito chimico e biologico. La laurea in biotecnologie mi ha aiutato parecchio in questo, ho seguito vari corsi interessanti come chimica, chimica organica, biochimica, enzimologia, ingegneria proteica, ecc.
Ma ovviamente non bastano da sole queste competenze, serve anche padronanza in altri ambiti, tra i quali si può citare quello chimico-fisico, matematico e computazionale. Infine, anche se non è una vera e propria competenza, ma un requisito essenziale, serve tanta tanta passione per fare un lavoro del genere.
“Nel 2017 hai fondato 3D Protein Imaging. Cos’è di preciso e come questo progetto si interseca con la tua passione di illustratore?”
Finita la magistrale ho iniziato a riflettere su come impiegare le mie competenze al meglio e mi sono anche accorto che molte persone in laboratorio avevano necessità di fare delle immagini di strutture proteiche che poi avrebbero utilizzato per presentazioni, tesi e articoli. I software che vengono utilizzati attualmente per generare le illustrazioni molecolari sono molto potenti, ma hanno un fondamentale difetto: è molto difficile imparare ad usarli al meglio.
Da questo presupposto nasce l’idea di sviluppare un’applicazione online che sia semplice da usare, ma che permetta agli utenti che la utilizzano di produrre loro stessi immagini di strutture molecolari di alta qualità in maniera semplice e rapida. L’applicazione, che si chiama Protein Imager, è liberamente accessibile al sito www.3dproteinimaging.com/protein-imager.
Protein Imager è anche uno strumento didattico molto utile per i giovani che approcciano al mondo della biologia strutturale per la primissima volta.
“Grazie Gianluca Tomasello per aver dedicato il tuo prezioso tempo per l’intervista con microbiologiaitalia.it. Prima di salutarci vuoi aggiungere qualcos’altro?”
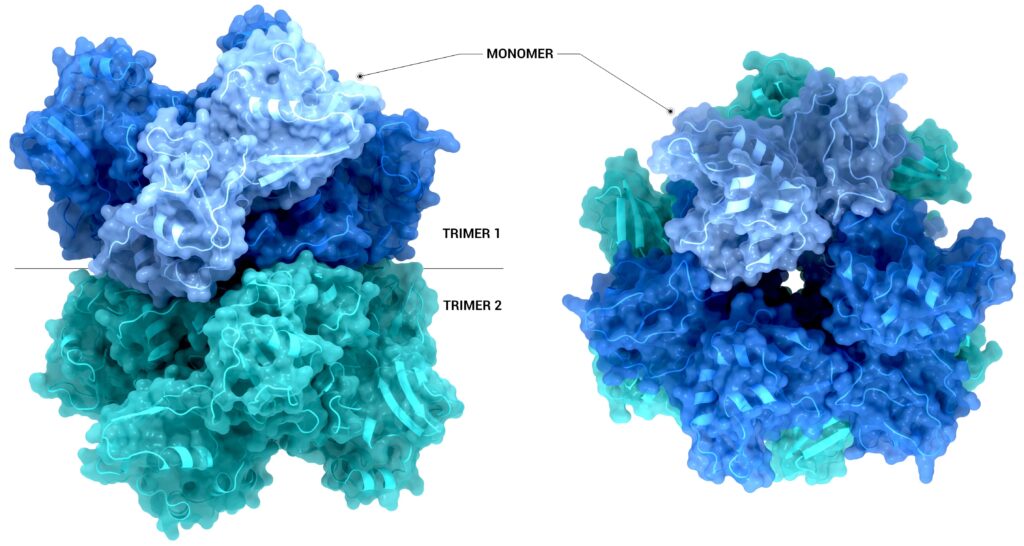
Grazie a voi! Vorrei concludere mostrando alcuni esempi di illustrazioni proteiche che ho avuto il piacere di fare recentemente per la Coronavirus Structural Task Force di Amburgo (www.insidecorona.net) per una special issue sull’intero proteoma del coronavirus.