Il microvirus ϕX174 è un batteriofago appartenente alla famiglia dei Microviridiae. Sono definiti microvirus per il loro genoma molto piccolo, compreso fra 4.5 e 6kb. ϕX174 in particolare, ha avuto un ruolo molto importante nello sviluppo della biologia molecolare.
Storia del microvirus ϕX174
Tra il 1920 ed il 1930 molti batteriofagi vennero scoperti, tra cui quelli appartenenti alla famiglia dei microvirus. Il più famoso di questa famiglia è proprio ϕX174. Robert Sinsheimer, riconoscendo le proprietà uniche di questo virus, iniziò a studiarlo verso la fine del 1950, e scoprì che contrariamente a tutti i genomi dei fagi scoperti fino a quel momento, quello del microvirus ϕX174 era a singolo filamento di DNA. Nel 1978 Sanger e altri suoi colleghi furono i primi a determinare la sequenza nucleotidica dell’intero genoma di un organismo, e fu proprio quella di ϕX174. Scoprirono così che molti dei geni del microvirus ϕX174 si sovrapponevano, mettendo in discussione il concetto di gene fino ad allora conosciuto.

Genoma di ϕX174
Infatti, 7 geni su 11 di ϕX174 risiedono in una regione con estese “reading frames” sovrapposte che coprono più di metà genoma del microvirus. Questi 7 geni di ϕX174 codificano per proteine non strutturali, mentre i rimanenti 4 geni (denominati J, F, G, H) codificano per proteine strutturali di ϕX174 e non si sovrappongo fra loro. L’uso di “reading frames” sovrapposte serve per aumentare la mole di informazione genetica codificata da un genoma piccolo come quello del microvirus ϕX174.
Il gene A di ϕX174 per esempio, si sovrappone ai geni A+, B e K. La proteina codificata da A ha un ruolo cruciale nella replicazione di ϕX174 ed interagisce quindi con numerose proteine dell’ospite. Inoltre è in grado di coevolvere con l’ospite, per cui mutazioni adattive su questo gene possono contemporanemante anche creare mutazioni nei geni sovrapposti modificandone le proprietà. Per questo, i geni A+ e K codificano per proteine non essenziali nella replicazione di ϕX174, mentre il gene B è in grado di tollerare numerose mutazioni senza significative alterazioni funzionali.
Allo stesso modo il gene E, che si sovrappone al fondamentale gene D, facilita la lisi cellulare, per cui non ha un ruolo cruciale nella replicazione ma velocizza il processo di rilascio del microvirus ϕX174.

Replicazione nell’ospite di ϕX174
I microvirus dipendono dall’RNA polimerasi della cellula ospite per poter sintetizzare il proprio mRNA. Tuttavia l’RNA polimerasi ha bisogno di DNA a doppio filamento, per cui i microvirus devono trasformare il proprio DNA a singolo filamento positivo (+) in uno a doppio prima di poter procedere con la trascrizione dei geni virali. Questa versione a doppio filamento viene definita “DNA in forma replicativa“, e viene sintetizzata durante la Fase I di replicazione del DNA di ϕX174, usando solo proteine dell’ospite.
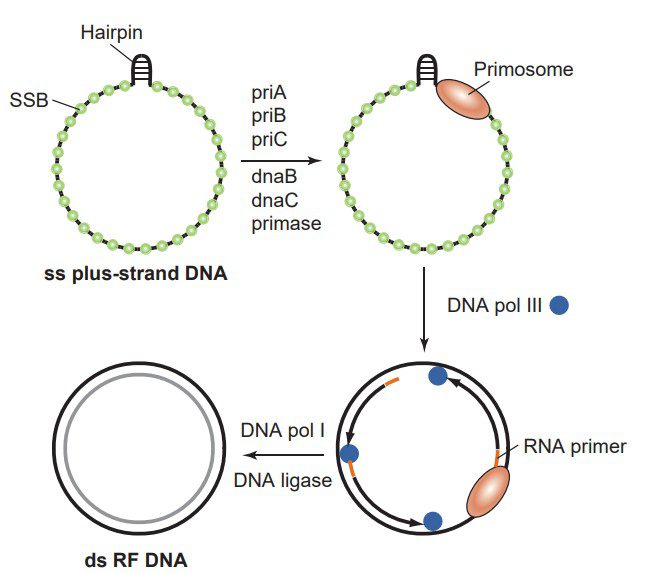
Tuttavia la singola molecola di DNA a doppio filamento non è sufficiente a generare tutte e 100 le “particelle” fagiche normalmente prodotte durante l’infezione di questo microvirus. La Fase II della replicazione di ϕX174 si occupa di amplificare il numero di molecole di DNA in forma replicativa, fino a 10 per cellula. Questo processo avviene tramite due fasi distinte (Figura 4):
- Nella prima fase si forma il DNA a singolo filamento (+) del microvirus, tramite un meccanismo chiamato “rolling circle“. Il filamento (+) di ϕX174 viene tagliato in un sito specifico dalla proteina virale A (1), e tramite un’elicasi si stacca dal filamento (-). Come il filamento (+) di ϕX174 si stacca, viene subito legato dalla proteine SSB (2), ed in contemporanea la DNA polimerasi III dell’ospite estende l’estremità 3′ libera (2) e sintetizza un nuovo filamento (+) di DNA di ϕX174. Una volta completato il cerchio viene staccato (4) il singolo filamento (+) dal completo DNA a doppio filamento in forma replicativa.
- Nella seconda fase viene sintetizzata (5), tramite un complesso di sette proteine dell’ospite, definito primosoma, una nuova molecola di DNA a doppio filamento di ϕX174, a partire dal singolo filamento (+) del microvirus.

Impacchettamento del DNA a singolo filamento
Biosintesi del genoma ed impacchettamento nel procapside di ϕX174 avvengono in contemporanea. Infatti nella fase III della replicazione del microvirus, la proteina virale C ridireziona il complesso di replicazione (proteina A, ds DNA, proteina rep, e la DNA polimerasi III). Per cui, anziché rilasciare il filamento (+) libero di ϕX174 per una nuova sintesi, il complesso si associa ad un nuovo procapside del microvirus, ed inserisce il singolo filamento (+) al suo interno. A questo punto, proteina virale J si occupa di legare il DNA da impacchettare e lo conduce dentro il procapside del microvirus. Questo complesso di procapside + DNA viene chiamato provirione. Dopo che avviene la lisi dalla cellula, con la dissociazione della proteina D, questo prende il nome di virione.
Bibliografia
- Fundamentals of Molecular Virology, 2nd edition, Acheson
- Kirchberger PC, Martinez ZA, Ochman H. Organizing the Global Diversity of Microviruses. mBio. 2022 Jun 28;13(3):e0058822. doi: 10.1128/mbio.00588-22. Epub 2022 May 2. PMID: 35491833; PMCID: PMC9239249.
- Doore SM, Baird CD; 2012 University of Arizona Virology Undergraduate Lab; Roznowski AP, Fane BA. The evolution of genes within genes and the control of DNA replication in microviruses. Mol Biol Evol. 2014 Jun;31(6):1421-31. doi: 10.1093/molbev/msu089. Epub 2014 Mar 4. PMID: 24600050.
- Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, Hutchison CA, Slocombe PM, Smith M. Nucleotide sequence of bacteriophage phi X174 DNA. Nature. 1977 Feb 24;265(5596):687-95. doi: 10.1038/265687a0. PMID: 870828.