Felix M. Key e il suo team hanno ricostruito la storia di una sierovariante di Salmonella enterica che infetta in maniera specifica l’uomo dai resti umani recuperati nell’Eurasia occidentale. I resti (ben 2739!) sono stati analizzati tramite l’uso di una nuova tecnica di screening batterica denominata HOPS che ha permesso di esaminarne i genomi dei batteri presenti al di sopra.
Questo ci dimostra quanto la paleomicrobiologia abbia ancora tanto da offrirci.
Salmonella paratyphi C
Il genere Salmonella è costituita da batteri Gram-negativi. Sono importanti agenti patogeni per l’uomo e per gli animali; negli esseri umani la maggior parte causa una gastroenterite autolimitante. Tuttavia, S. typhi e S. paratyphi A, B e C causano febbre enterica nell’uomo.
S.paratyphi C fu isolato da un uomo in Francia nel 1988. Non condivide un antenato comune con altri agenti che causano il tifo e sembra essersi discostato da un antenato comune con S. choleraesuis (un patogeno suino) e da allora ha accumulato diversità genetica nel processo di adattamento all’uomo.
La salmonella che infettava sia animali che uomini
I genomi recuperati dai resti archeologici, ha permesso di ricostruire otto antichi genomi di Salmonella enterica, alcuni di questi risalenti a 6.500 anni fa. Sei sono progenitori di Salmonella paratyphi C, un ceppo che raramente infetta l’uomo. Tuttavia è interessante evidenziare che la Salmonella ritrovata nei resti archeologici fosse un progenitore della S. paratyphi C e infettava sia gli uomini che gli animali. La specializzazione di questo ceppo nell’infezione dell’uomo potrebbe essere avvenuta durante il periodo del neolitico.
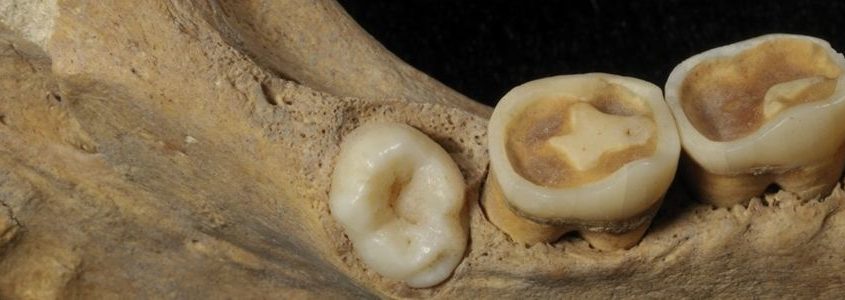
Neolitico, un passaggio cruciale
Secondo lo studio pubblicato su Nature Ecology & Evolution, l’adattamento sarebbe avvenuto durante il Neolitico, quando i nostri antenati iniziarono ad abbandonare le pratiche della caccia ed iniziarono a svilupparsi l’allevamento e l’agricoltura.
L’allevamento ha permesso inoltre di aumentare le possibilità di infezione da parte della Salmonella dovuta alla maggiore vicinanza delle feci animali agli uomini.
Prospettive future
Il genoma di un agente patogeno è difficile da ricostruire perché è difficile che l’organismo umano ne conservi i segni dell’infezione. Il risultato ottenuto permetterà di ricostruire la storia evolutiva di alcuni degli agenti patogeni presenti ancora oggi grazie all’uso delle nuove tecnologie.
Fonti
- “Emergence of human-adapted Salmonella enterica is linked to the Neolithization process” Felix M. Key, Cosimo Posth, Johannes Krause Nature Ecology & Evolution
- https://www.epicentro.iss.it/salmonella/
1 commento su “La pelomicrobiologia stupisce ancora: Salmonella e neolitico”