Topi knock-out
I modelli animali sono sempre più importanti per lo studio e la comprensione di particolari meccanismi biologici che avvengono nel nostro organismo. Tra i modelli animali maggiormente utilizzati, anche per lo studio di patologie umane, ci sono i modelli murini. Questo è dovuto alle elevate somiglianze genetiche e fisiopatologiche che ci sono tra uomo e topo (Figura 1).
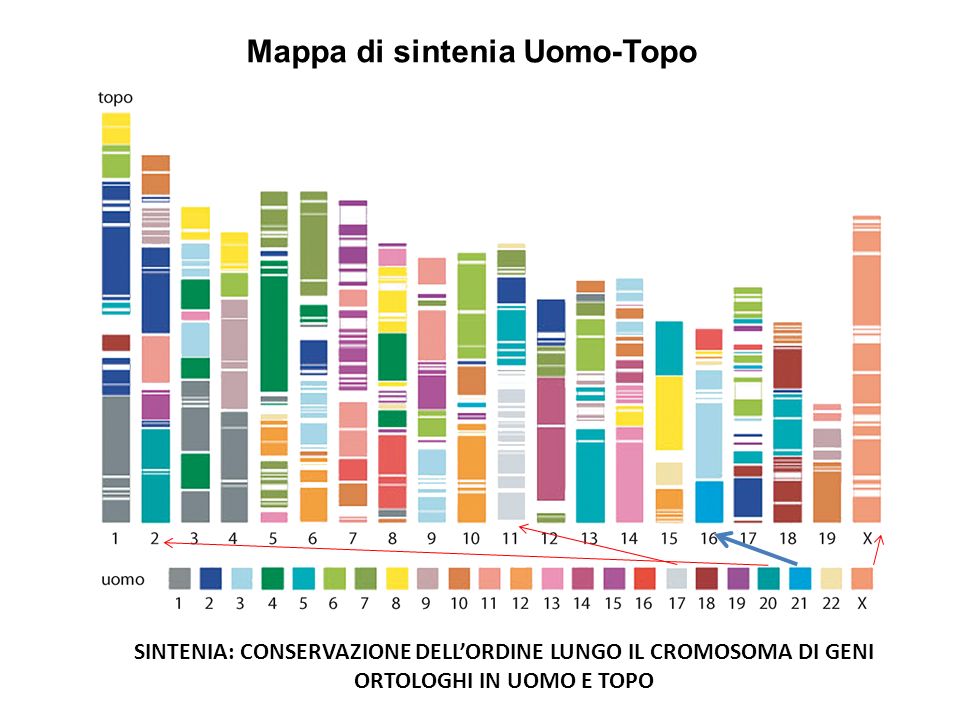
Da questo è derivata la necessità di creare topi geneticamente modificati, in cui uno o più geni vengono silenziati: i topi knock-out. Il primo topo knock-out è stato creato negli anni Ottanta da Mario Capecchi, Martin Evans e Oliver Smithies, che, per questo, ricevettero il Premio Nobel per la Medicina nel 2007.
Possiamo distinguere due tipi di topi knock-out:
- Topi knock-out convenzionali: in cui il gene è silenziato già a livello embrionale. Questi modelli murini presentano, però, delle problematiche. Spesso, infatti, il silenziamento precoce di alcuni geni risultava incompatibile con la vita; altre volte, invece, si attuava un meccanismo di compensazione che impediva di visualizzare gli effetti del silenziamento del gene di interesse;
- Topi knock-out condizionali: in cui il gene viene silenziato in un determinato momento della vita dell’animale (controllo temporale) o solamente in un dato tessuto/cellula (controllo spaziale).
In questo articolo ci occuperemo del secondo caso, spiegando la procedura attraverso cui è possibile ottenere dei knock-out condizionali. Il sistema maggiormente utilizzato è il sistema Cre-loxP.
Il sistema Cre-loxP
Il primo articolo scientifico che presenta il sistema Cre-loxP è stato pubblicato da K. Rajewsky nel 1995 sulla rivista Science. Da allora è diventato il metodo di elezione per la creazione di topi knock-out condizionali e, di conseguenza, per lo studio delle patologie umane.
Che cosa è?
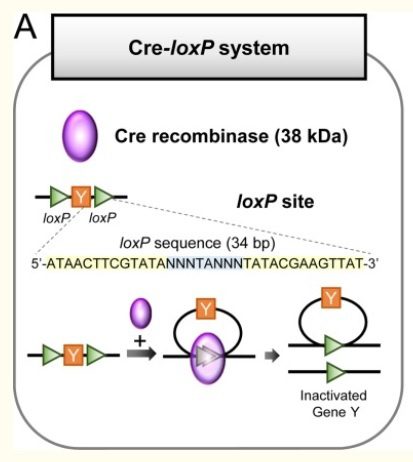
Il sistema si basa su una metodica di ricombinazione tipica del batteriofago P1. I due elementi importanti della procedura sono:
- Cre: è una DNA ricombinasi di 38 kDa, prodotta dal gene cre (ciclizzazione ricombinasi);
- loxP (locus di crossover del fago P1): una sequenza di 34 pb, costituita da due sequenze palindromiche di 13 pb ciascuna e una regione centrale di 8 pb.
La ricombinasi Cre può essere considerata una forbice molecolare che riconosce i siti loxP e taglia in corrispondenza di questi, che generalmente fiancheggiano un gene di interesse (Figura 2). A seconda dell’orientamento dei siti loxP si può avere escissione, inversione o traslocazione del gene fiancheggiato (floxed). Generalmente la metodica è utilizzata per l’escissione – quindi, l’inattivazione – del gene di interesse, per cui i siti loxp presentano orientamento diretto.
Come funziona?
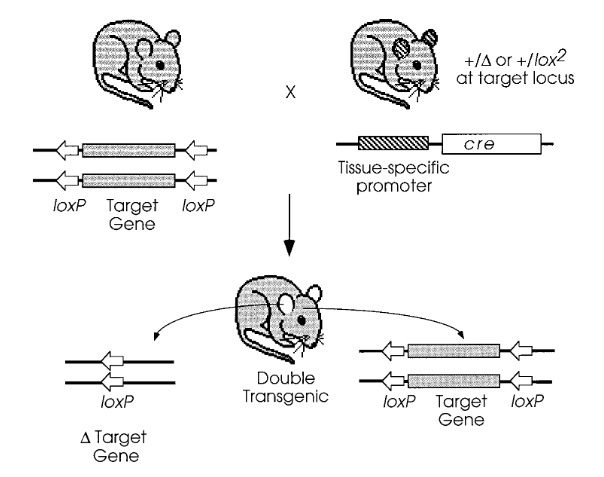
Prima di procedere con la creazione dei topi knock-out condizionali, è necessario capire come è fatto il gene di interesse e, quindi, come modificarlo. Sicuramente i siti loxP devono fiancheggiare quelle porzioni del gene, che risultano fondamentali per la sua espressione: gli esoni. Dopo aver fatto questo bisogna generare un allele target, a livello del quale sono presenti i siti loxP, che sarà utilizzato per creare un ceppo di topo loxP. Questo verrà fatto incrociare con un ceppo di topo che esprime la ricombinasi Cre. Dall’incrocio si otterrà un topo doppio-transgenico, che presenterà sia il gene cre che il gene floxed. Il riconoscimento, da parte della ricombinasi Cre, dei siti loxP, porterà all’eliminazione di quel gene (Figura 3).
Il sistema cre-loxP e il controllo spazio-temporale
L’importanza di questo sistema sta nella possibilità di controllare da un punto di vista spaziale e/o temporale l’espressione della ricombinasi Cre e il silenziamento del gene di interesse. Questo è possibile perché si va ad agire sugli elementi regolatori della trascrizione: promotori ed enhancer.
Per quanto riguarda il controllo spaziale, è possibile silenziare un gene solo a livello di un dato tessuto o, addirittura, solo a livello di un dato citotipo cellulare. Per fare questo è necessario porre il gene cre sotto il controllo di un promotore specifico per un dato tessuto/cellula (Figura 3). Così, la ricombinasi Cre sarà espressa solo in un determinato sito e solo qui riconoscerà i siti loxP e silenzierà il gene.
Da un punto di vista temporale, il controllo è mediato dall’utilizzo di sistemi inducibili, cioè l’espressione della ricombinasi sarà mediata da induttori esogeni. Quelli maggiormente utilizzati sono il tamoxifene e la tetraciclina.
Il sistema Cre-loxP inducibile: tamoxifene e tetraciclina
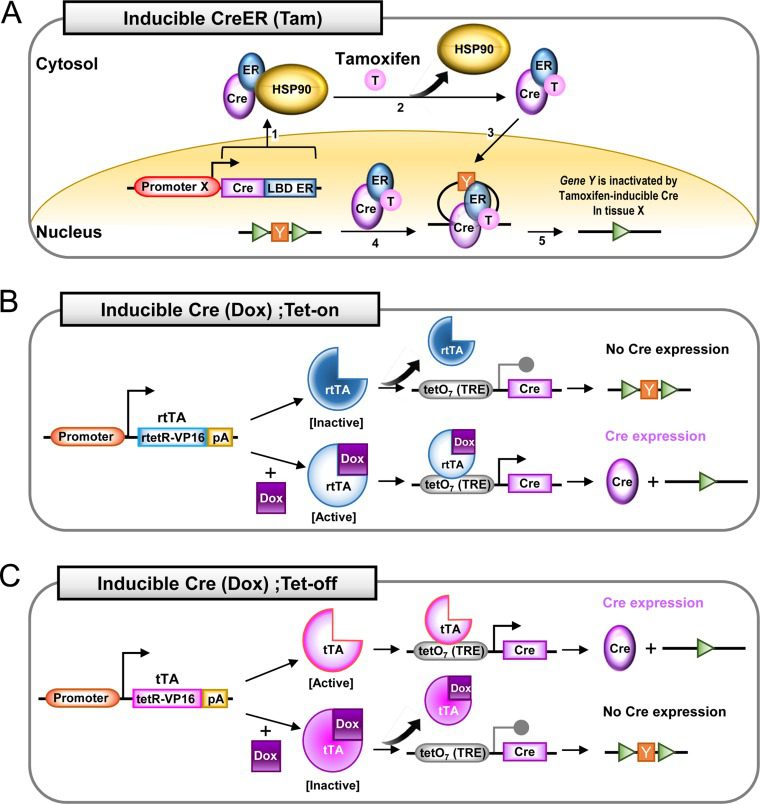
Se si vuole utilizzare come induttore esogeno il tamoxifene (chemioterapico agonista degli estrogeni), è necessario creare una proteina di fusione, la proteina Cre-ERT (Figura 4). Questa sarà costituita da ricombinasi Cre e il dominio di legame al ligando (LBD) mutato del recettore per gli estrogeni. La presenza di un LBD mutato impedisce il legame di estrogeni e/o progesterone endogeni. Nel momento in cui è presente il ligando sintetico (il tamoxifene), somministrato, mediante iniezione intraperitoneale, questo lega il recettore e permette alla proteina di fusione di traslocare nel nucleo, dove svolge la sua funzione.
Un altro sistema inducibile è quello basato sulla tetraciclina, o meglio su un suo derivato, la doxiciclina, (Figura 4) somministrata attraverso il mangime e/o l’acqua. Questo sistema è costituito da tre elementi:
- la proteina rtTA: il transattivatore controllato dalla tetraciclina in senso inverso;
- la proteina tTA: il transattivatore controllato dalla tetraciclina;
- TRE: l’elemento responsivo alla tetraciclina (noto anche come operone della tetraciclina, TetO) che controlla l’espressione della ricombinasi.
Inoltre, questo sistema è disponibile in due modalità:
- Tet-on: la doxiciclina lega rtTA, che può, quindi, legare l’operone e attiva la trascrizione del gene cre;
- Tet-off: la doxiciclina lega tTA, questa non può legarsi all’operone, a cui è legata normalmente, e inibisce l’espressione della ricombinasi. In questo sistema, la doxiciclina viene somministrata attraverso il mangime o l’acqua.
Conclusione
Essendo questo sistema largamente utilizzato, ad oggi sono state sviluppate numerose linee di modelli animali contenenti la ricombinasi Cre, che sono condivise dai ricercatori mediante networks internazionali. Non solo, si sta cercando di implementare la metodica per migliorarne l’efficienza, in modo da ottenere uno studio sempre più accurato della fisiopatologia umana.
Emanuela Pasculli
Fonti
- “Il Nobel che mise KO i geni. Intervista a Mario Capecchi (1 Ottobre 2012). Estrapolato da https://archivioscienze.scuola.zanichelli.it/in-evidenza/2012/10/01/mario-capecchi-un-nobel-che-mette-i-geni-ko/;
- Kim, Hyeonhui, et al. “Mouse Cre-LoxP system: general principles to determine tissue-specific roles of target genes.” Laboratory animal research 34.4 (2018): 147-159;
- Sauer, Brian. “Inducible gene targeting in mice using the Cre/loxsystem.” Methods 14.4 (1998): 381-392;
- Kuhn, Ralf, et al. “Inducible gene targeting in mice.” Science 269.5229 (1995): 1427-1429;
- Nagy, Andras. “Cre recombinase: the universal reagent for genome tailoring.” genesis 26.2 (2000): 99-109;
- Branda, Catherine S., and Susan M. Dymecki. “Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice.” Developmental cell 6.1 (2004): 7-28.