Come già noto, all’indomani dell’emergenza sanitaria relativa alla malattia da nuovo coronavirus (Sars-CoV2, meglio conosciuto come Covid-19), i ricercatori di tutto il mondo hanno unito le loro forze nella progettazione e nella formulazione di un vaccino anti-Covid 19, che dovrebbe essere pronto per il 2021.
A distanza di alcuni mesi, tutti gli studi convergono verso un vaccino basato sulle sequenze antigeniche delle proteine strutturali S, le cosiddette Spike Protein, che sono foriere di una risposta immunitaria efficace e duratura.
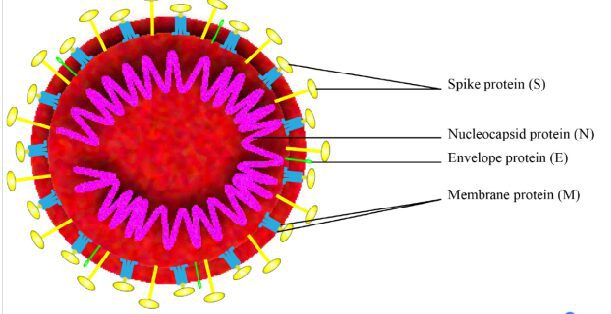
Figura 1 – Struttura completa del Covid-19
Ruolo delle proteine S nella patogenesi da Covid 19
Le spike protein sono glicoproteine di superficie che mediano l’attacco del virus all’enzima ACE (Angiotensyn Converting Enzyme) e la sua fusione per endocitosi con la cellula ospite.
Più in dettaglio, le spike protein presentano due sub unità: la sub unità S1, che contiene il dominio di legame al recettore (Receptor Binding Domain-RBD) a livello dell’estremità C-terminale, si lega al recettore ACE sulla cellula ospite, mentre la sub unità S2 effettua la vera e propria fusione, grazie ad un peptide di fusione della membrana interna (FP), due ripetizioni a 7 peptidi (HR), una membrana prossimale esterna regione (MPER) e un dominio trans-membrana (TM) .
Nelle figure sottostanti, la cinetica delle spike protein, descritta da Mc Lellan e collaboratori, attraverso una tecnica spettroscopica, denominata Cryo- EM.
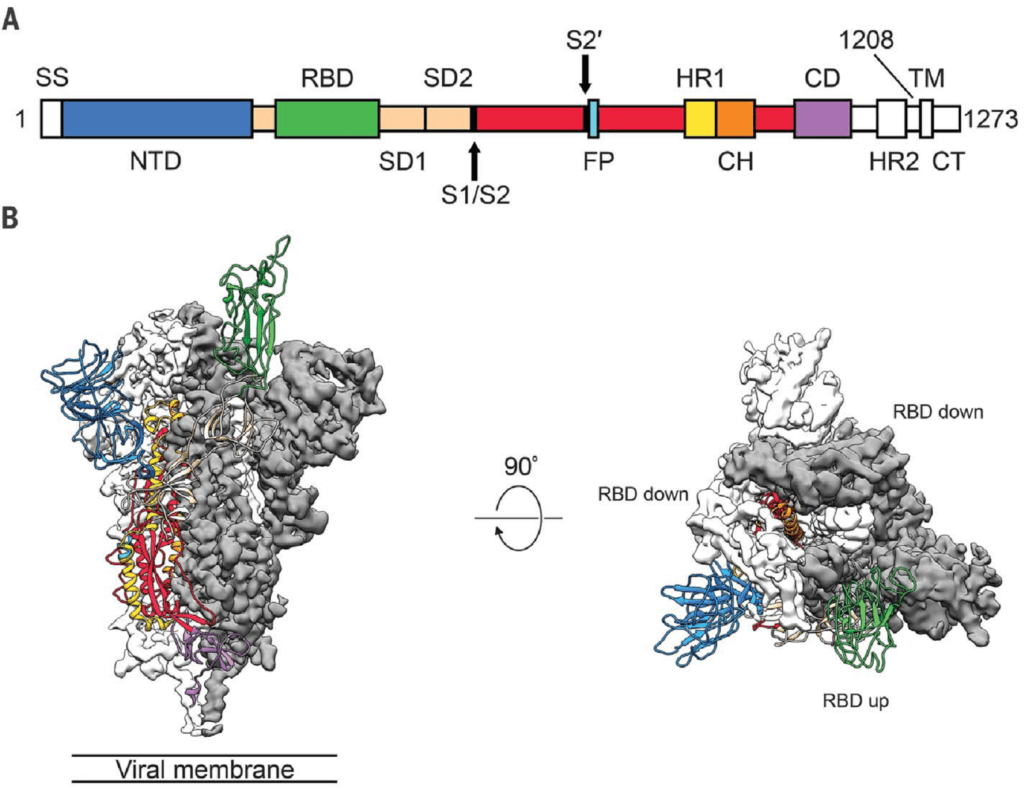
Figura 2 – A) Struttura lineare della proteina S; B)Movimento “a cerniera” dell’Receptor Binding Protein”
La proteina S è un trimero che segue un meccanismo di fusione definito di classe I: presenta, infatti, una conformazione pre- fusione metastabile e subisce un sostanziale riarrangiamento strutturale per fondere la membrana virale con la membrana della ospite. Lo stato prefusione è definito “down” ed è quello di non accessibilità al recettore ACE. In esso uno dei tre domini RBD della sub unità S1 è rivolto verso l’alto (colore verde, in figura). Quando la sub unità S1 si lega al recettore, effettua un movimento a cerniera, che espone o nasconde gli altri due RBD (vedi figura) e fa assumere alla sub unità S2 una conformazione post fusione ancor meno stabile. Questo stato è definito “up” perché la membrana virale può fondere con la cellula.
La risposta del sistema immunitario
Le spike protein stimolano sia la risposta immunitaria cellulo mediata, sia la risposta immunitaria anticorpale.
La prima risposta, ricordiamo, si deve alla differenziazione dei linfociti T in CD4 helper e CD8+ citotossici, che rispettivamente riconoscono l’antigene proteico presente sulla superficie cellulare e lo fagocitano. La seconda si deve alla differenziazione dei linfociti B in plasmacellule che producono anticorpi neutralizzanti, sotto lo stimolo della stessa risposta cellulo mediata.
Precedenti studi sul Sars-CoV hanno dimostrato che la risposta immunitaria umorale è risultata di più breve durata nei pazienti in convalescenza, mentre la risposta cellulo-mediata garantisce una protezione più a lungo termine, anche fino ad 11 anni dall’infezione.
Tra le proteine del virus SARS presenti nelle cellule mononucleate del sangue periferico dei pazienti convalescenti, la risposta cellulo-mediata contro le proteine strutturali è risultata essere la più immunogenica rispetto a quella contro le proteine non strutturate. In particolare, ha prevalso la risposta cellulo-mediata contro le proteine strutturali N e S, risultata quella di più lunga durata.
Un esperimento in vitro condotto da Wang e colleghi ha inoltre dimostrato che le spike protein di Sars-CoV2 mediano l’ingresso del virus anche nei linfociti T, pur presentando questi ultimi un più basso livello di espressione dell’enzima ACE. Dallo stesso esperimento è emerso inoltre che i linfociti T sono molto più sensibili a Sars-CoV2 rispetto ai virus congeneri, Sars-CoV e Mers-Cov.
Tutte queste scoperte rafforzano l’ipotesi di un vaccino anti-Sars-CoV2 che stimoli più efficacemente la risposta immunitaria cellulo-mediata.
Sulla base di questa evidenza, Ahm e collaboratori, tutti afferenti all’Università di Hong Kong, hanno fornito, attraverso un approccio bio-informatico, una serie di epitopi cellulari per entrambe le risposte identici tra Sars-CoV e Sars-CoV2, e che pertanto potrebbero essere utilizzati come guida (lead) per futuri studi sperimentali.
Tali epitopi sono stati individuati tra quelli già risultati positivi ai saggi sperimentali, che nel caso della risposta cellulo-mediata sono il saggio immunoenzimatico ELISPOT, il saggio delle citochine e il saggio di legame al complesso maggiore di istocompatibilità.

Tra i 115 epitopi risultati positivi al saggio delle cellule T, 27 sono risultati totalmente identici in Sars-CoV e Sars-CoV2 e, di questi, 19 sono stati mostrati positivi al saggio di legame MHC con cinque alleli: HLAA *02:01; HLAB*40:01; HLA-DRA*01:01; HLA-DRB1*07:01; HLA-DRB1*04:01. Tra gli stessi 27 epitopi, 11 erano presenti sulla proteina S. Il tutto è riportato nella tabella seguente.

Per quanto riguarda, invece la risposta anticorpale, i ricercatori hanno riscontrato che gli epitopi lineari nella sub unità S1, la più esposta tra le due, non trovano molte somiglianze tra Sars-CoV e Sars-CoV2, il che non renderebbe possibile lo sviluppo di un vaccino in grado di proteggere da entrambe le specie virali. Gli stessi ricercatori rimandando ad ulteriori studi relativi ad un vaccino anticorpale basato sugli epitopi della subunità S2.
Stato dell’arte sul vaccino basato sulle Spike Protein
Un articolo di Zhang e colleghi, pubblicato il 29 Marzo scorso sulla rivista Vaccines prende in esame tutti i frammenti di proteina S utilizzabili come antigeni per lo sviluppo di un vaccino anti-covid 19: l’intera proteina S, il dominio RBD, la subunità S1, il dominio N-terminale NTD e FP.
- La proteina S per intero può fornire un più alto numero di epitopi riconoscibili dalle cellule T e, più in generale, una maggiore immunogenicità.
- Il dominio di legame RBD è quello più conservato all’interno della sub unità S1 ed inoltre è stato dimostrato sperimentalmente che esso contiene più epitopi che possano fungere da antigene.
- Il dominio N Terminale è il meno conservato.
Conclusioni
Le spike protein (proteine di superficie) sono il bersaglio più promettente per la messa a punto di un vaccino, perché giocano un ruolo chiave nella patogenesi del virus, ed in più perché sono riconosciute direttamente dal sistema immunitario.
La caratterizzazione strutturale pre-fusione delle spike protein di Sars-CoV2 apre la strada a numerose possibilità di ingegnerizzazione al fine di aumentarne l’antigenicità e per delineare la struttura di eventuali ulteriori strutture mutate dello stesso Sars-CoV2.
La somiglianza strutturale delle proteine S di Sars-CoV2 ai congeneri delle Beta Coronaviridae, Sars-Cov e Mers-CoV, fa si che un solo vaccino possa funzionare per tutte e tre le specie virali.
Adele D’Alessandro
Bibliografia
- Preliminary Identification of Potential Vaccine Targets for the COVID-19 Coronavirus (SARS-CoV- 2) Based on SARS-CoV Immunological Studies Syed Faraz Ahmed 1,†, Ahmed A. Quadeer 1,*,† and Matthew R. McKay 1,2; Viruses 2020, 12, 254
- Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation
- Daniel Wrapp1*, Nianshuang Wang1*, Kizzmekia S. Corbett2, Jory A. Goldsmith1, Ching-Lin Hsieh1, Olubukola Abiona2, Barney S. Graham2, Jason S. McLellan1† Science 367, 1260–1263 (2020)
- SARS-CoV-2 infects T lymphocytes through its spike protein-mediated membrane fusion
- Xinling Wang1, Wei Xu1, Gaowei Hu1, Shuai Xia1, Zhiping Sun1, Zezhong Liu1, Youhua Xie1, Rong Zhang1, Shibo Jiang1,2 and Lu Lu 1 Cellular & Molecular Immunology
- Progress and Prospects on Vaccine Development against SARS-CoV-2 Jinyong Zhang 1, Hao Zeng 1, Jiang Gu 1, Haibo Li 1, Lixin Zheng 2,* and Quanming Zou 1,* Vaccines 2020, 8, 153
inserire nel DNA di E.coli come per insulina la sequenza DNA della proteina spike ricavata dalla sequenza RNA Covid-19 ormai nota,
produzione di proteina spike a livello intestinale da E.coli modificati ingeriti.
risposta immune da sistema linfonodale intestinale Peyer.
valutazione di percentuali di curabilità per pz, sint0matici intestinali verso sintomatici polmonari
L’articolo di Wang et al è stato ritirato
Serietà vorrebbe che le conclusioni basate su questo articolo fossero modificate di conseguenza.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7136698/
This article has been retracted.
Retraction in: Cell Mol Immunol. 2020 July 10; : 1 See also: PMC Retraction Policy
SARS-CoV-2 infects T lymphocytes through its spike protein-mediated membrane fusion
Xinling Wang,#1 Wei Xu,#1 Gaowei Hu,#1 Shuai Xia,1 Zhiping Sun,1 Zezhong Liu,1 Youhua Xie,1 Rong Zhang,1 Shibo Jiang,corresponding author1,2 and Lu Lucorresponding author1
Buonasera,
La ringrazio per la precisazione. A breve un articolo sul fatto e le nuove conclusioni.
A presto
Un vaccino che agisca sull’angiotensina non puo’ interferire con il suo funzionamento e provocare problemi cardiaci o renali ?
Salve io ho fatto l esame spike rbd il risultato e 774,00 volevo sapere se era buono come risultato perché non c’è una tabella per poter confrontare grazie
How to choose a high quality protective equipment
http://www.bulletproof-equipment.com