Quale relazione tra RNA e SARS-COV-2?
Il COVID-19 (CoronaVirus Disease 19) è una malattia infettiva causata dall’agente virale SARS-CoV 2 (Severe Acute Respiratory Syndrome CoronaVirus 2) che ha scatenato la crisi sanitaria iniziata nel 2020 e divenuta pandemia. Questo scenario ha indotto il mondo scientifico ad una corsa alla ricerca di approcci diagnostici e terapeutici per fronteggiare l’emergenza. Tra gli innumerevoli studi, un capitolo all’avanguardia è sicuramente quello degli RNA non codificanti (ncRNA) come microRNA (miRNA), short interfering RNA (siRNA) e long non coding RNA (lncRNA) e la possibilità che questi influenzino la patogenesi virale, diventando automaticamente potenziali target farmacologici.
I ncRNA
Gli RNA non codificanti sono RNA che non vengono tradotti in proteine ma svolgono un ruolo regolatorio nei confronti di processi molecolari e cellulari. Per molto tempo gli RNA messaggeri (mRNA) hanno rappresentato l’unico tipo di RNA con ruolo significativo, al contrario di tutti gli altri RNA non codificanti considerati dei sottoprodotti privi di significato biologico chiaro. A seguito della scoperta del ruolo cruciale dell’RNA ribosomiale (rRNA) e dell’RNA transfer (tRNA), è nato un immediato interesse per tutta la classe di RNA non codificanti. Recentemente, miRNA, siRNA e lncRNA si sono rivelati inaspettatamente importanti per processi biologici, fisiologici e patologici relativi all’infezione da parte di SARS-CoV-2.
La relazione tra microRNA e SARS-CoV-2
I microRNA sono la classe prevalente dei piccoli RNA non codificanti e sono lunghi meno di 200 nt. Influenzano l’espressione genica cellulare attraverso il meccanismo di RNA interference, il quale entra in gioco anche quando la cellula subisce un’infezione virale. Il meccanismo prevede che ci sia un appaiamento tra il mRNA virale ed il miRNA ospite: se c’è perfetta complementarietà, il messaggero virale è degradato ma se l’appaiamento è imperfetto (è presente almeno un mismatch), il messaggero non è degradato ma la sua traduzione è inibita dal blocco dei ribosomi.
La regolazione dei microRNA
Le proteine più importanti per l’invasione cellulare da parte di SARS-CoV-2 (es. ACE2, TMPRSS2, ADAM17) sono regolate da alcuni miRNA. Un esempio è il microRNA let-7a-g/i che, attraverso il meccanismo di RNA interference, è capace di sotto-regolare l’espressione della proteina TMPRSS2, contrastando l’avanzata del virus nell’ospite. Let-7a-g/i potrebbe spiegare la disparità di genere nella gravità della COVID-19 che si associa ad una condizione patologica grave maggiormente nell’uomo rispetto alla donna.
Diversità ormonale
Alla base di questo dato statistico c’è la diversa condizione ormonale, in quanto nella donna gli estrogeni stimolano gli RNA della famiglia let-7, i quali intensificano il meccanismo inibitorio nei confronti di proteine come TMPRSS2, sottraendo al virus la possibilità di avanzare. Al contrario, gli androgeni non garantiscono lo stesso meccanismo di difesa nell’uomo perché let-7 non è presente in grandi quantità. Quindi, la stimolazione di questo miRNA potrebbe rappresentare un bersaglio terapeutico strategico per evitare i risvolti gravi della COVID-19 negli individui.
La relazione tra lncRNA e SARS-CoV-2
I long non coding RNA sono una classe di RNA non codificanti lunghi più di 200 nucleotidi. Sono molecole regolatrici coinvolte nelle interazioni virus-ospite e nell’infiammazione tissutale. Infatti, alcuni lncRNA regolano l’espressione dei geni stimolatori dell’interferone (ISGS). E’ risaputo che l’interferone è la prima linea di difesa del nostro organismo contro un’infezione virale, quindi alcuni lncRNA possono allertare l’ospite che sta subendo l’infezione.
Un esempio di questa relazione è NEAT1, un RNA non codificante che, quando carente, comporta una riduzione della risposta infiammatoria nell’individuo affetto da SARS-CoV-2. Inoltre, altri studi dimostrano come questi RNA modulino anche l’infezione a carico dei neuroni, essendo il SARS-CoV-2 un virus neurotropico. I lncRNA vicini ad alveoli e bulbo olfattivo possono aumentare l’espressione di citochine (es. IL-2, IL-7, TNF-α etc.) scatenando la tempesta citochinica che è responsabile della perdita della percezione olfattiva.
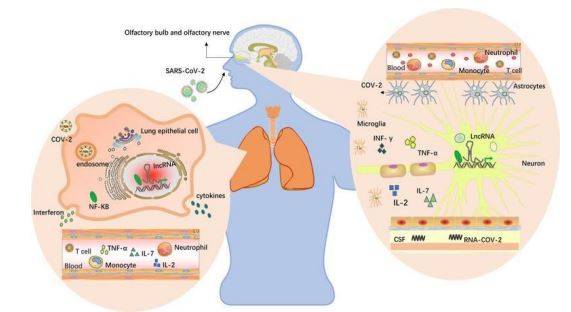
infezione nasale, raggiunge il SNC attraverso il bulbo olfattivo. A questo livello, i lncRNA possono mediare la trascrizione di geni coinvolti nella tempesta citochinica. Analogamente, a sinistra è raffigurata la stessa attività dei lncRNA nelle cellule epiteliali dei polmoni infette.
La scoperta delle relazioni tra lncRNA e il SARS-CoV-2 ha portato alla luce uno studio svolto sul virus della rabbia che, essendo anch’esso neurotropico, può essere applicato anche in questo caso: attraverso un complesso meccanismo di modificazione del DNA della cellula ospite, il lncRNA EDAL può promuovere l’espressione del polipeptide antivirale PCP4L1 che contrasta l’avanzamento del virus.
La relazione tra siRNA e SARS-CoV-2
I short interfering RNA (siRNA) sono piccoli ncRNA a doppio filamento, lunghi circa 21-23 bp. Anche questi, come i miRNA, proteggono gli organismi dalle infezioni virali mediante il meccanismo di RNA interference. Al contrario dei microRNA, questi agiscono solo in organismi come piante ed invertebrati. Nonostante ciò, questa classe di RNA non codificanti sta dimostrando un potenziale terapeutico interessante che consiste nell’indurre il meccanismo di RNA interference nelle cellule di mammifero mediante l’utilizzo di siRNA esogeni specifici.
Progettare nuovi siRNA
L’idea è quella di progettare siRNA in grado di bersagliare specifiche regioni del genoma di SARS-CoV-2 o RNA subgenomici come quelli codificanti per la proteina o altre, in modo da contrastare l’avanzamento virale. La somministrazione dei siRNA esogeni avverrebbe per via sistemica o locale e il modo migliore per il rilascio in cellula si è dimostrato essere l’utilizzo dei nanovettori polimerici. L’aspetto più affascinante di questo potenziale terapeutico è la possibilità di progettare siRNA contro specifiche sequenze che fanno la differenza nel genoma virale delle varianti.

Oggi, infatti, la sfida del mondo scientifico è quella di fronteggiare le varianti emergenti che insorgono numerose a causa dell’accumulo di mutazioni del genoma virale di SARS-CoV-2. Quest’ultimo, avendo un genoma a RNA, replica attraverso una RNA polimerasi RNA dipendente che è priva di attività di proof-reading, caratteristica che spiega la facilità con cui si susseguono le mutazioni. Al contrario di SARS-CoV-2, infatti, i virus a DNA replicano mediante la DNA polimerasi che oltre a garantire la sintesi dell’acido nucleico, svolge un’attività di “controllo” e “pulizia” durante la replicazione, abbattendo di gran lunga il numero di mutazioni.
I ncRNA: una classe di RNA per combattere SARS-CoV-2
Tra le moltissime varianti di SARS-CoV-2, indichiamo con VOC (Variants Of Concern) le cinque che destano maggiore preoccupazione per la salute pubblica: Alfa (Regno Unito, Dicembre 2020), Beta (Sud Africa, Dicembre 2020), Gamma (Brasile, gennaio 2021), Delta (India, dicembre 2020), Omicron (Sud Africa, novembre 2021).
Le molteplici relazioni appena dimostrate tra il mondo degli RNA non codificanti ed il SARS-CoV-2 con le sue varianti, ci fanno sperare che, approfondendo la ricerca in merito, il potenziale ruolo diagnostico, prognostico e predittivo di questi RNA possa concretizzarsi ed essere un’arma in più contro la pandemia della COVID-19.
Fonti
- Henzinger H, Barth DA, Klec C, Pichler M. Non-Coding RNAs and SARS-Related Coronaviruses. Viruses. 2020;12(12):1374. Published 2020 Dec 1. doi:10.3390/v12121374
- Kadam SB, Sukhramani GS, Bishnoi P, Pable AA, Barvkar VT. SARS-CoV-2, the pandemic coronavirus: Molecular and structural insights. J Basic Microbiol. 2021;61(3):180-202. doi:10.1002/jobm.202000537
- Kim D, Lee JY, Yang JS, Kim JW, Kim VN, Chang H. The Architecture of SARS- CoV-2 Transcriptome. Cell. 2020;181(4):914-921.e10. doi:10.1016/j.cell.2020.04.011
- Naqvi AAT, Fatima K, Mohammad T, et al. Insights into SARS-CoV-2 genome, structure, evolution, pathogenesis and therapies: Structural genomics approach.Biochim Biophys Acta Mol Basis Dis. 2020;1866(10):165878. doi:10.1016/j.bbadis.2020.165878
- Zhang P, Wu W, Chen Q, Chen M. Non-Coding RNAs and their Integrated Networks. J Integr Bioinform. 2019;16(3):20190027. Published 2019 Jul 13. doi:10.1515/jib-2019-0027
- Narozna M, Rubi ̇ śB. Anti-SARS-CoV-2 Strategies and the Potential Role of miRNA in the Assessment of COVID-19 Morbidity, Recurrence, and Therapy. Int J Mol Sci. 2021;22(16):8663. Published 2021 Aug 12. doi:10.3390/ijms22168663
- Sardar R, Satish D, Gupta D. Identification of Novel SARS-CoV-2 Drug Targets by Host MicroRNAs and Transcription Factors Co-regulatory Interaction Network Analysis. Front Genet. 2020;11:571274. Published 2020 Oct 14. doi:10.3389/fgene.2020.571274
- Yang Q, Lin F, Wang Y, Zeng M, Luo M. Long Noncoding RNAs as Emerging Regulators of COVID-19. Front Immunol. 2021;12:700184. Published 2021 Aug 2.
- doi:10.3389/fimmu.2021.700184
- Sui B, Chen D, Liu W, et al. A novel antiviral lncRNA, EDAL, shields a T309 O- GlcNAcylation site to promote EZH2 lysosomal degradation. Genome Biol. 2020;21(1):228. Published 2020 Sep 1. doi:10.1186/s13059-020-02150-9
- Mehta A, Michler T, Merkel OM. siRNA Therapeutics against Respiratory Viral Infections What Have We Learned for Potential COVID-19 Therapies?.Adv Healthc Mater. 2021;10(7):e2001650. doi:10.1002/adhm.202001650