Introduzione: cenni storici
L’acido ribonucleico o RNA è un acido nucleico coinvolto nei processi fondamentali delle nostre cellule e di tutte le cellule presenti in natura. È inoltre anche la prima molecola comparsa sulla Terra ad aver determinato l’inizio della vita.
Il primo step che condusse alla scoperta delle molecole di RNA avvenne intorno all’anno 1870, quando il chimico Friedrich Miescher isolò dai globuli bianchi la nucleina. Con il tempo però, la nucleina si rivelò essere un secondo acido nucleico, il DNA, che nel 1952 venne confermato essere la molecola detentrice dell’informazione biologica. Sempre negli stessi anni veniva ipotizzata l’idea di un’altra molecola, l’RNA, capace di trasferire l’informazione biologica dalla molecola di DNA alle proteine. In particolare, nel 1954, Crick avanzava l’ipotesi dell’adattatore al “RNA Tie Club”, fondato proprio con lo scopo di svelare i segreti dietro alla molecola di RNA.
Successivamente, negli anni ’60, vennero isolati sia l’mRNA che il tRNA, molecole chiavi nel processo di sintesi proteica.
Struttura RNA
Per capire la struttura dell’RNA possiamo immaginare una collana di perle, composta quindi da un unico filamento formato da più unità che si susseguono. Ogni perla corrisponde ad un nucleotide, che, legandosi ad altri nucleotidi, forma l’acido ribonucleico.
A sua volta un nucleotide è formato da più elementi: uno zucchero, uno o più gruppi fosfato e una base azotata. Nel caso dell’RNA, il ribosio (zucchero) si lega in posizione 5’ ad un gruppo fosfato e in posizione 1’ ad una base azotata. Le basi azotate sono invece di quattro tipi: adenina, timina, guanina e uracile.
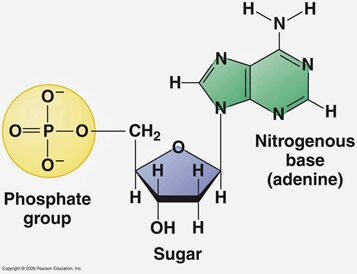
Il legame che unisce due nucleotidi è il legame fosfodiesterico che vede quindi coinvolto l’ossidrile legato al carbonio 3 del primo nucleotide e il fosfato legato al carbonio 5 del secondo nucleotide. Per convenzione la polarità della molecola sarà 5’->3′.
Struttura tridimensionale
A differenza del DNA, l’RNA può ripiegarsi su sé stesso per formare, in analogia con le proteine, delle strutture secondarie e terziarie. La struttura tridimensionale è fondamentale per il ruolo biologico di molte molecole di RNA e si forma dall’appaiamento tra le basi azotate complementari presenti nella sequenza nucleotidica.
L’esempio più eclatante di struttura tridimensionale è la struttura del tRNA (Fig. 2) la cui funzione, come vedremo in seguito, è quella di fungere da adattatore.
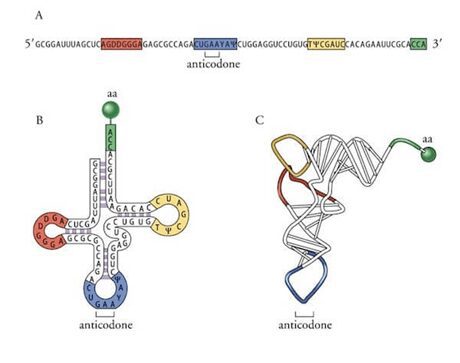
Nella Figura 2 è possibile osservare come il tRNA si ripiega per formare legami tra le basi azotate dei nucleotidi anche distanti e quali nucleotidi sono coinvolti nella formazione dei legami intramolecolari.
Funzioni
Nonostante la struttura di base delle molecole di RNA sia sempre la stessa, molecole diverse con dimensioni diverse possono avere funzioni completamente differenti. Tra le funzioni più importanti si hanno:
- Trasporto dell’informazione;
- Storage dell’informazione;
- Regolazione;
- Catalisi.
Trasporto dell’informazione
La prima funzione è sicuramente quella più conosciuta: le molecole di RNA permettono il trasporto dell’informazione dal DNA alle proteine. Questa funzione si esplica in particolare grazie all’intervento di tre tipi di RNA: mRNA, tRNA e rRNA.
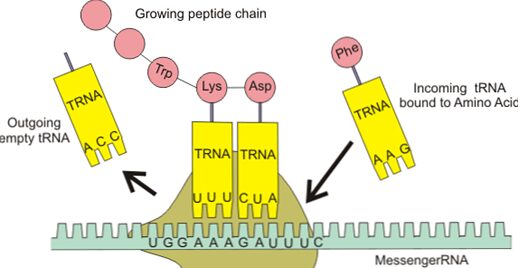
L’mRNA si forma tramite il processo di trascrizione dei geni, che consiste, come lascia intendere la parola stessa, nella trascrizione dell’informazione dal DNA all’RNA. A questo punto, l’mRNA forma un complesso insieme ad un ribosoma, formato a sua volta da proteine e rRNA. Infine, il tRNA funge da adattatore: da una parte infatti lega gli amminoacidi che formeranno la catena polipeptidica della proteina, dall’altra riconosce unità presenti nell’mRNA chiamate codoni. Questo concetto è alla base della traduzione, processo che permetterà di sintetizzare la catena polipeptidica (Fig. 3).
Storage dell’informazione
Sappiamo che, nelle nostre cellule, il DNA detiene l’informazione biologica. Questo però non è vero per tutti gli organismi viventi. Virus come il Coronavirus utilizzano infatti come materiale genetico l’RNA. Quest’ultimo può essere sia a doppio filamento (in analogia con il DNA) sia a singolo filamento. Tra i virus con un RNA a singolo filamento si distinguono i virus con un genoma a polarità negativa e virus con genoma a polarità positiva. Il genoma con una polarità positiva avrà lo stesso orientamento dell’mRNA, che come abbiamo visto è essenziale per poter effettuare la sintesi proteica. Il genoma con polarità negativa è invece complementare all’mRNA. Anche in questo caso comunque si può ricavare il filamento di mRNA.
Infine, un caso curioso è l’esistenza dei cosiddetti retrovirus, virus che pur avendo un genoma a RNA possono, mediante l’enzima trascrittasi inversa, convertirlo in DNA (Fig. 4).

Regolazione
Su questo punto non ci soffermeremo molto. Basti sapere che, oltre alle tre molecole di RNA principali (mRNA, tRNA, rRNA), esistono molte altre molecole di dimensioni molto variabili che contribuiscono alla regolazione dell’espressione genica. Tra queste ritroviamo ad esempio miRNA, siRNA e IncRNA.
Catalisi
La funzione catalitica assume una notevole importanza soprattutto se si pensa alle origini della vita. Come abbiamo già accennato, infatti, le prime molecole ad essere presenti nel cosiddetto brodo primordiale erano molecole di RNA. Affinché potesse esistere un ambiente formato da solo RNA queste dovevano assumere anche il ruolo catalitico associato solitamente alle proteine. Queste molecole di RNA, i ribozimi, esistono tutt’ora, basti pensare agli rRNA presenti nei ribosomi che contribuiscono alla sintesi proteica mediante un meccanismo catalitico oltre che strutturale.
Maturazione dell’RNA
Con maturazione dell’RNA ci riferiamo a quel processo presente nelle cellule eucariotiche che porta alla formazione di una molecola di RNA matura a partire da una molecola di pre-RNA.
In questo paragrafo ci soffermeremo in particolare sulla maturazione degli mRNA. Essa ha un duplice scopo: proteggere e permettere il riconoscimento dell’RNA attraverso la modificazione chimica delle sue estremità (capping e poliadenilazione) e eliminare le sequenze introniche (splicing). Una sequenza intronica è una sequenza che si interpone tra due sequenze codificanti.
Capping
Il capping è una modificazione chimica all’estremità 5’ della molecola. Nello specifico consiste nell’aggiunta di un nuovo nucleotide che viene successivamente metilato (7-metilguanosina). Le tappe della reazione sono le seguenti:
- Una RNA trifosfatasi agisce sul nucleotide all’estremità 5’ rimuovendo il fosfato γ (l’ultimo fosfato);
- Una guanilil trasferasi aggiunge un GMP (guanosina mono-fosfato) attraverso la formazione di un legame trifosfato 5’-5’ (diverso dal classico legame fosfodiesterico);
- Aggiunta del metile in posizione 7 al nucleotide appena aggiunto.
Il capping è la prima modificazione chimica apportata all’RNA e avviene in contemporanea alla trascrizione: l’RNA nascente viene subito modificato affinché possa essere protetto dall’attività delle nucleasi. Oltre alla funzione di protezione, il capping aumenta l’efficienza della traduzione, facilita il trasporto dell’ RNA fuori dal nucleo e aumenta l’efficienza dello splicing.
Poliadenilazione
La prima modifica apportata all’RNA è quindi l’aggiunta di un “cappuccio” che corrisponde ad un nuovo nucleotide metilato. Che succede invece quando la trascrizione finisce e si forma una seconda estremità? A questo punto attraverso il processo di poliadenilazione viene aggiunta una coda di poli-adenine che può contare fino a 200-250 adenine.
L’intero processo si svolge in due fasi: fase di inizio e fase di allungamento. Nella fase di inizio interviene l’enzima PAP che aggiunge poche adenine, dopodichè nella fase di allungamento oltre all’enzima PAP interviene PABPN1 con il quale aggiunge rapidamente le restanti.
Le funzioni della poliadenilazione sono simili a quelle del capping: protezione contro la degradazione, aumento di efficienza della traduzione, favorire lo splicing e la traslocazione nel citoplasma.
Splicing
L’ultimo processo che analizzeremo è forse quello più interessante e consiste nell’eliminazione delle sequenze introniche. Questo tipo di sequenze non codificano per alcuna proteina e si trovano interposte tra le sequenze codificanti di tutto il genoma.
Il processo di splicing in realtà si può operare in vari modi, ma quello più diffuso è lo splicing nucleare che riguarda i precursori degli mRNA. Nello splicing nucleare è fondamentale la presenza di un grande complesso chiamato spliceosoma formato a sua volta da RNA e proteine. Il suo compito durante il processo è sia quello di riconoscere i giusti siti dell’RNA sia operare l’avvicinamento delle sequenze da unire.
Per finire, è interessante conoscere il significato di splicing alternativo. Lo splicing alternativo è un processo che permette di partire da un unico trascritto primario di RNA per arrivare a due o più trascritti diversi di RNA maturo. In questo modo uno stesso gene può arrivare a codificare per due o più proteine diverse.
Fonti
- Brock, Thomas D., et al. (2016) Biologia dei microrganismi : microbiologia generale, ambientale e industriale. 14. ed, Pearson Italia;
- Amaldi, F., Benedetti, P., Pesole, G., & Plevani, P. (2011). Biologia Molecolare. Milano : C.E.A. Casa Editrice Ambrosiana.