Cosa sono i ribosomi
Il processo biologico, definito traduzione, il quale permette l’espressione dell’mRNA in proteine, avviene mediante complessi macromolecolari: i ribosomi (figura 1).
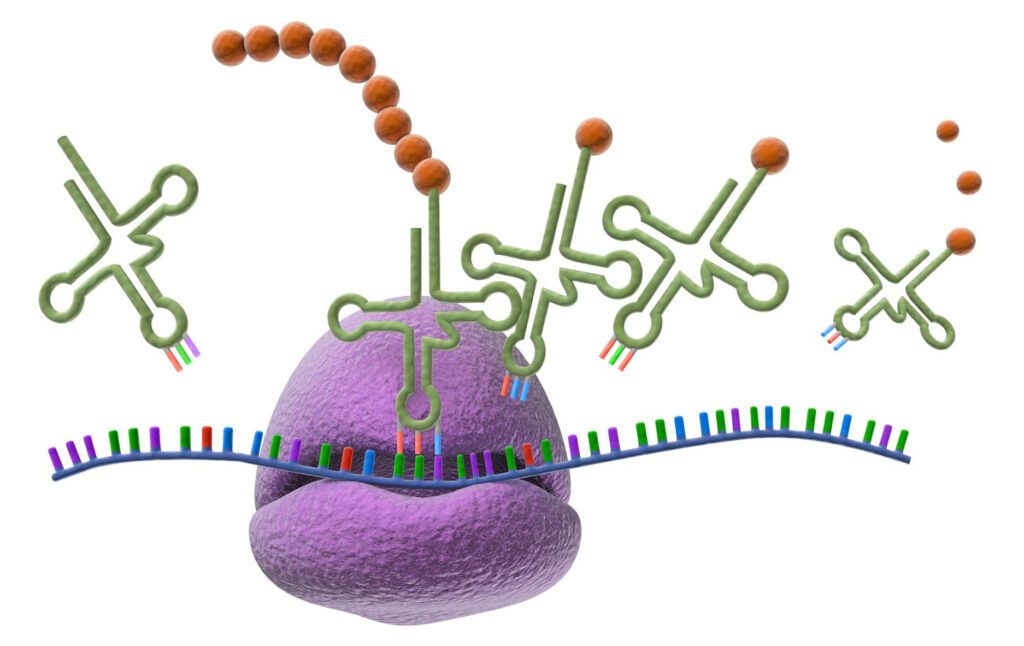
I ribosomi sono stati scoperti al microscopio nel 1953 dal biologo rumeno George Emil Palade, sono caratterizzati da due macromolecole RNA ribosomiali (rRNA) e proteine. Le proteine e gli RNA ribosomiali sono aggregati mediante legami non covalenti, primariamente da legami idrogeno.
Ma dove si trovano i ribosomi? Tali strutture le ritroviamo nel citoplasma di tutte le cellule, sia procariotiche che eucariotiche. Nei procarioti, gli organelli in esame sono liberi, poichè non presente alcun reticolo endoplasmatico che possa legarli.
Ribosomi struttura
Le due subunità ribosomiali asimmetriche, osservate al microscopio si mostrano strutturalmente molto simili. La frazione maggiore ha una forma di barca con un’estremità più appuntita, mentre la frazione minore è formata da una testa e un corpo legati da un collo più sottile.
I ribosomi batterici dalla massa di circa 2700 kDa e un diametro di 20nm, hanno il coefficiente di sedimentazione è 70S e le due frazioni che costituiscono il complesso hanno rispettivamente 30S e 50S. Nel dettaglio la subunità 30S è costituita da 21 proteine differenti e da una molecole di rRNA 16S. Invece, la frazione di 50S è formata da 34 proteine e due molecole di RNA (5S e 23S). A differenza dei ribosomi eucarioti che hanno una massa di 4000kDa e mostrano un coefficiente di sedimentazione totale di 80S e due subunità rispettivamente da 60S (45 proteine e 3 molecole di rRNA) e 40S (33 proteine e una moleca di rRNA 18S).
Cos’è il coefficiente di sedimentazione?
Ricordiamo che il coefficiente di sedimentazione, indicato con la lettera S, in unità Svedberg, si riferisce alla velocità di sedimentazione nella centrifugazione a ultravelocità. Ad incidere sul parametro sono un insieme di fattori come peso molecolare, grandezza, peso e forma. Per queste motivazioni la somma delle due subunità (30S + 50S) non corrisponde al coefficiente totale di 70S.
Una regione piana (piattaforma) nella lieve curvatura della subunità minore, forma un solco in corrispondenza con la depressione della subunità maggiore. Il solco tra le due subunità, accoglierà l’mRNA durante la traduzione. Nella frazione più grande del ribosoma sono presenti tre siti di legami per le molecole di tRNA:
- A: sito di attacco, in cui l’anticodone del tRNA si lega al codone dell’mRNA, ponendo il proprio aminoacido affianco alla catena polipeptidica che si ta formando;
- P (sito polipeptidico): il tRNA cede il proprio aminoacido alla catena in formazione, nel sito di condensazione;
- E (sito Exit): il tRNA dopo aver ceduto il proprio aminoacido, si posiziona nel sito di distacco, prima di lasciare il ribosoma e ricominciare il ciclo.
Ribosomi funzione
Come ben si può capire, la funzione dei ribosomi fondamentale è quella di supporto agli RNA nella sintesi proteica. Ricordiamo le fasi della traduzione procariotica ed eucariotica, che si susseguono schematicamente (figura 2):
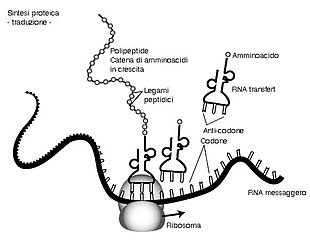
Formazione del complesso d’inizio
L’rRNA presente nella subunità minore del ribosoma, si lega al sito di legame situato vicino l’estremità 5′ sull’mRNA. Il legame tra il ribosoma e l’mRNA è facilitata, nei procarioti, dalle sequenze di Shine-Dalgarno, basi presenti tra 5 e 10 nucleotidi prima del codone di avvio. L’anticodone complementare del tRNA, caricato con metionina formilata a livello dell’azoto (fMet) o metionina nel caso degli eucarioti, si lega al codone iniziale AUG dell’mRNA. Successivamente la frazione maggiore si lega al complesso d’inizio costituito dal tRNA caricato dell’aminoacido unito alla subunità minore e mRNA. Il tRNA con fMet/Met passa la sito P e nel sito A si posiziona il secondo codone dell’mRNA. Ogni tRNA è specifico per un singolo aminoacido, l’ossidrile libero in 3′ del braccio accettore del tRNA è il punto di aggancio dell’aminoacido.
Nella prima fase della traduzione, la principale differenza tra procarioti ed eucarioti riguarda i fattori d’inizio. I fattori d’inizio dei procarioti permettono il legame di tutti i componenti, svolgono attività per la preparazione del ribosoma: IF1 allontana le subunità 30S e 50S, IF2 promuove il legame del complesso fMet-tRNA al ribosoma 30S e IF3 lega l’mRNA alla subunità 30S. Negli eucarioti, dobbiamo ricordare che l’mRNA presenta un CAP ed una forcina da trattare con più fattori d’inizio (EIF4F, EIF2, EIF2B, EIF3, EIF6, EIF5, EIF1 e EIF1A) per agevolare il ribosoma nel proprio lavoro.
Allungamento
Nel sito P, perso il legame con la formilmetionina, si lega il secondo aminoacido caricato sul tRNA situato nel sito A. La frazione maggiore del ribosoma catalizza il legame peptidico tra i due aminoacidi, quindi l’rRNA funziona da ribozima. Dal sito P, il tRNA senza aminoacido passa al sito E e si stacca. Il ribosoma procede in direzione 5′-3′ e procede di un codone, in modo che il complesso tRNA-polipeptide si trovi nel sito P. Nei procarioti, i fattori EF-1A, EF-1B e EF-2 intervengono in questa fase di allungamento. EF-1A porta il tRNA successivo al sito A del ribosoma, EF-1B funge da fattore di scambio di nucleotidi rimpiazzando la molecola di GDP risultante con un nuovo GTP e l’ultimo fattore EF-2 media la traslocazione. Mentre negli eucarioti, eEF-1 porta l’aminoacil tRNA successivo al sito A del ribosoma e eEF-2 media interviene nella traslocazione.
Terminazione
L’ultima fase avviene quando l’enzima incontra i codoni di stop (UAA, UAG e UGA). Ai codoni di stop non si lega nessun tRNA ma fattori di rilascio. Nei procarioti RF1 riconosce i codoni UUA e UAG, mentre RF2 UAA e UGA ed il loro legame al ribosoma è coadiuvato da RF3 (proteina GTP-dipendente). Negli eucarioti i fattori di rilascio sono due, eRF1 riconosce i tre codoni di terminazione e eRF3 coadiuva il distacco del polipeptide dal ribosoma.
Fonti
- Biologia, microbiologia e tecnologie di controllo sanitario – Fabio fanti, Zanichelli
- https://www.treccani.it/enciclopedia/
- Immagine in evidenzia : Proteinsyntesen
Crediti immagini
- Immagine di copertina: https://ndla.no
- Figura 1: https://ndla.no
- Figura 2: https://it.wikipedia.org/wiki/Sintesi_proteica