Introduzione allo splicing
La sequenza codificante di un gene è costituita da una serie di codoni a 3 nucleotidi che specificano per un singolo aminoacido della catena polipeptidica prodotta durante il processo di traduzione. Contrariamente dai batteri e dai fagi, in cui la sequenza codificante è continua, per i geni degli eucarioti le sequenze codificanti (esoni) vengono interrotte periodicamente da segmenti di sequenze non codificanti (introni). Esiste, inoltre, una certa variabilità sia nella dimensione che nel numero degli introni che compongono i differenti geni: sebbene possano essere anche corti, solitamente sono più lunghi degli esoni e possono arrivare fino a 800.000 nucleotidi. In seguito alla trascrizione di un RNA primario, gli introni devono essere rimossi e gli esoni legati per creare l’mRNA maturo di quel gene specifico. Tale processo prende il nome di splicing dell’RNA.
La chimica dello splicing
Gli introni presentano specifiche sequenze conservate necessarie all’attuazione del processo di splicing. In particolare, l’estremità 5’ dell’introne, detto anche sito donatore, presenta la sequenza GU mentre l’estremità 3’ o sito accettore presenta la sequenza AG. Fondamentale per il processo di splicing è anche una terza sequenza completamente all’interno dell’introne: il cosiddetto punto di ramificazione in corrispondenza del quale si trova un’adenina conservata con un residuo libero OH in posizione 2’.
La prima reazione di transesterificazione (chiamata così poiché la A di ramificazione, partecipa a tre legami fosfodiesterici) è avviata dal 2’OH dell’adenina. Questo gruppo effettua un attacco nucleofilo sul gruppo fosfato della guanina conservata del sito donatore al 5’. Si rompe così il legame fosfodiesterico tra zucchero e fosfato alla giunzione introne-esone e l’estremità 5’ libera dell’introne viene unita alla A del punto di ramificazione formandosi una struttura ad “ansa”. A questo punto, il residuo OH al 3’ dell’esone rimasto libero esercita un altro attacco nucleofilo sul gruppo fosfato presente all’estremità 3’ dell’introne e si forma una struttura definita “a cappio” e contemporaneamente le due sequenze codificanti vengono “legate” (Fig. 1). Per la chimica di questo processo non vi è consumo di energia poiché si verificano due reazioni accoppiate in cui vi è solo uno scambio di legami.
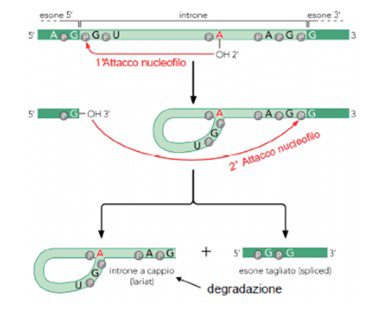
Lo splicing dell’RNA è mediato dallo spliceosoma
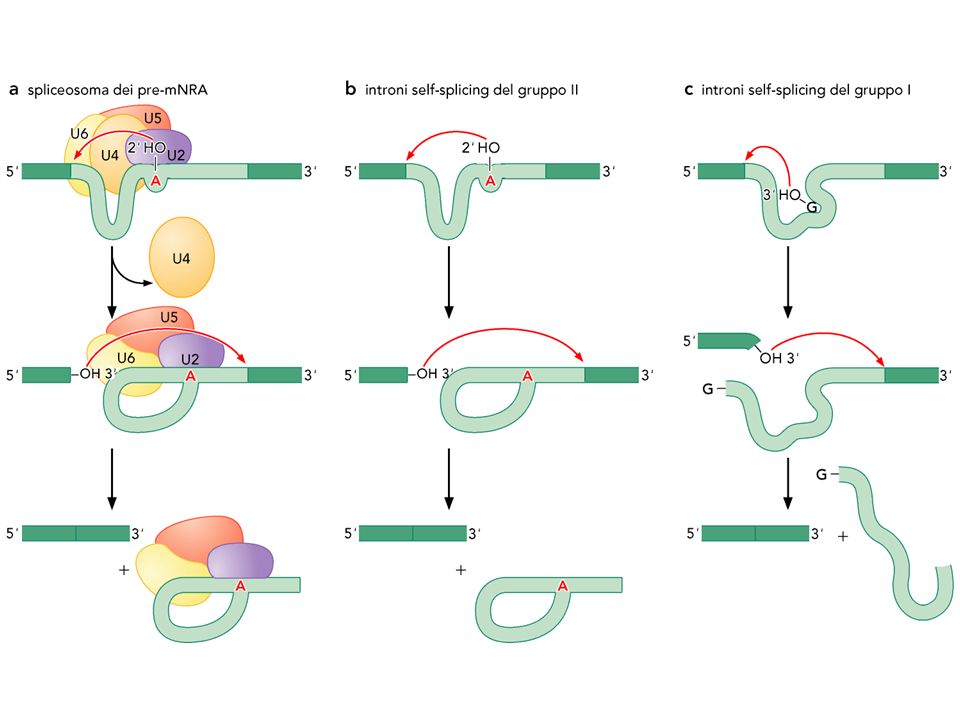
Le reazioni di transesterificazione sono mediate da un complesso multi-enzimatico che prende il nome di spliceosoma costituito da 150 proteine e da 5 piccoli RNA nucleari o anche detti snRNA (U1, U2, U4, U5 e U6). Ciascuno di questi RNA è lungo dai 100 ai 300 nucleotidi e forma complessi con diverse proteine che prendono il nome di piccole ribo-nucleoproteine nucleari (snRNP). Tali complessi svolgono tre ruoli fondamentali nel processo di splicing:
- Riconoscere il sito donatore (estremità 5’) dell’introne e il punto di ramificazione;
- Favorire all’occorrenza la vicinanza di tali siti;
- Catalizzare il taglio e la giunzione dell’RNA.
In dettaglio:
- SnRNP U1 si associa all’estremità 5′ dell’introne
- SnRNP U2 è complementare alla regione centrale dell’introne comprendente la A di ramificazione;
- SnRNP U4 e U6 si associano, interponendosi nella regione tra U1 e U2 creando una distorsione dell’introne stesso (cappio);
- Dopo la rimozione del fattore U4, snRNP U6 si associa ad U2 esercitando un’attività catalitica che consente la liberazione dell’esone 1 e la formazione della struttura a cappio;
- SnRNP U5 a sua volta avvicina le estremità 3′ e 5′ degli esoni in modo da favorire la giunzione.

Lo splicing dell’mRNA senza spliceosoma mediato dagli introni self-splicing
Il termine self-splicing si riferisce all’ auto-capacità degli introni di rimuoversi dall’RNA in assenza di proteine in vitro.
Si distinguono:
- Introni self-splicing del gruppo I
- Introni self-splicing del gruppo II
Tuttavia, questi introni non sono da considerarsi veri enzimi poiché hanno un numero di turnover pari ad 1 (ovvero sono in grado di mediare solo un ciclo di maturazione dell’RNA) ma possono, in vitro, essere convertiti in ribozimi: molecole di RNA in grado di catalizzare una reazione chimica similmente agli enzimi.
Introni self-splicing del gruppo I
Affinché la reazione possa avvenire è necessario che la struttura degli introni di gruppo I abbia una “tasca” in grado di legare un nucleotide o un nucleoside presentante il ribosio. Inoltre, deve contenere una sequenza di guida interna per favorire l’appaiamento della stessa sequenza al sito di splicing 5’. In questo modo viene determinato il sito specifico dove avverrà il primo attacco nucleofilo. Gli introni di gruppo I, anziché un residuo A nel punto di ramificazione, utilizzano un nucleoside G con un residuo OH libero che determina un attacco nucleofilo in corrispondenza dell’estremità 5’ dell’introne. La seconda reazione, poi, prevede che l’estremità 3’ libero dell’esone si attacchi al 3’ dell’introne. I due esoni si legano e l’introne viene rilasciato in forma lineare invece che a “cappio” (Fig. 3).
Introni self-splicing del gruppo II
Il meccanismo di reazione nel caso degli intorni di gruppo II è essenzialmente lo stesso dello spliceosoma. L’introne utilizza il residuo di A nel punto di ramificazione per attaccare il legame fosfodiesterico al sito di splicing 5’. Questa reazione porta alla formazione di un cappio e, successivamente, il residuo 3’-OH dell’esone appena liberato esercita un attacco nucleofilo al sito di splicing 3’. A questo punto, l’introne viene rilasciato come cappio e i due esoni vengono assemblati (Fig. 3).
Lo splicing alternativo
Negli eucarioti è possibile che, da uno stesso gene, si possano ottenere differenti prodotti proteici (isoforme). Tale processo è mediato dallo splicing alternativo e si esplica in modo da assicurare che i differenti prodotti proteici siano espressi in differenti tipi cellulari. Ad esempio, da un unico pre-trascritto di mRNA contente 4 esoni, si possono generare due molecole di mRNA mature. I due messaggeri conterranno 3 esoni per ciascuno: due in comune e un terzo specifico per ciascuna isoforma (Fig. 4). In alcuni casi gli esoni possono essere anche estesi, saltati oppure alcuni introni possono essere anche mantenuti nell’RNA maturo.

L’editing dell’RNA
Come lo splicing, anche l’editing può modificare la sequenza di un RNA dopo il processo di trascrizione. Tale processo prevede però un cambiamento dell’informazione codificante dell’RNA a seguito di inserzione, delezione o sostituzione di singole basi.
L’editing si esplica attraverso due principali meccanismi:
- Deaminazione sito-specifica;
- L’inserzione o delezione di un’uridina dall’RNA guida.
Completato il processo di trascrizione, l’mRNA maturo subirà il processo di traduzione. Tale meccanismo è regolato dal codice genetico mediante il quale si ha la conversione della sequenza nucleotidica delle basi costituenti il DNA in sequenza amminoacidica costituente la catena polipeptidica nascente.
Deaminazione sito-specifica
In particolare, nel processo di deaminazione sito-specifica si verifica un cambiamento nella sequenza di uno specifico codone. L’enzima deaminasi, presente specificatamente in alcuni organi o tessuti, determina una conversione di una citosina in uracile per esempio, causando cambiamenti nella sequenza e, di conseguenza, nella lettura dell’mRNA. La proteina di nuova sintesi può quindi essere molto diversa nella composizione degli amminoacidi da quella prevista dal codice genetico.
L’inserzione e la delezione delle uridine ad opera degli RNA guida
La sequenza dell’RNA guida o gRNA presenta tre regioni fondamentali:
- Una prima regione definita “ancora” all’estremità 5’ che permette l’appaiamento complementare del gRNA alla regione dell’mRNA che deve subire l’editing;
- Una seconda regione che determina il sito specifico di inserzione delle U sul mRNA (processo catalizzato dall’enzima uridiltransferasi 3’ terminale);
- Una terza regione che presenta un segmento di poli-U all’estremità 3’ che trattiene il gRNA sulla sequenza dell’mRNA ricca in purine.
Il gRNA e l’mRNA formano un duplex RNA-RNA creando un’ansa a singola elica dal lato opposto rispetto all’inserzione delle U. Successivamente, un’endonucleasi taglia l’mRNA in corrispondenza del sito d’inserzione delle U e l’uridiltransferasi trasferirà le uridine nell’apertura. Infine, le due estremità dell’mRNA vengono riassemblate dall’RNA ligasi.
Fonti
- RNA Splicing | Learn Science at Scitable (nature.com)
- A self-splicing RNA excises an intron lariat – ScienceDirect
- RNA editing | FEMS Microbiology Reviews | Oxford Academic (oup.com)
Crediti immagini
- Immagine in evidenza: https://www.science.org/do/10.1126/science.adc9663/abs/_20220511_on_rna.jpg
- Figura 1: https://qph.cf2.quoracdn.net/main-qimg-a638b222ccf97a4dd05dfe03dd6f3905
- Figura 2: https://upload.wikimedia.org/wikipedia/commons/3/30/Spliceosome_ball_cycle_new2.jpg
- Figura 3: https://images.slideplayer.it/1/544427/slides/slide_28.jpg
- Figura 4: https://rgmedia.raportuldegarda.ro/2020/01/splicing-alternativ.jpg