Il pirosequenziamento è una tecnologia per il sequenziamento di DNA basato sulla sintesi (sequencing-by-synthesis, SBS). Questa tecnologia è basata sul lavoro pioneristico del Dr. Pål Nyrén del 1987. Il pirosequenziamento monitora la sintesi di DNA in tempo reale attraverso la bioluminescenza. Ogni nucleotide incorporato crea un segnale luminoso che viene registrato e convertito in sequenza nucleotidica.
Il pirosequenziamento fa parte del sequenziamento di nuova generazione (Next generation sequencing) che, in breve, prevede i seguenti passaggi:
- frammentazione del DNA da sequenziare;
- amplificazione attraverso la reazione a catena della polimerasi (polymerase chain reaction, PCR);
- sequenziamento.
Le applicazioni
Le principali applicazioni del pirosequenziamento sono:
- la ricerca di mutazioni in sequenze sconosciute, cioè il ri-sequenziando di pochi punti d’interesse;
- la genotipizzazione di batteri attraverso il sequenziamento del gene 16S rRNA;
- la genotipizzazione di virus per la ricerca di mutazioni che conferiscono farmaco-resistenza;
- la genotipizzazione di SNP (polimorfismi a singolo nucleotide);
- il sequenziamento di EST (expressed sequence tag) per studio dell’espressione genica e identificazione di nuovi trascritti.
Principio
Le reazioni del pirosequenziamento
Il pirosequenziamento è basato sulla sintesi, quindi il filamento di DNA da analizzare funge da stampo per il frammento di Klenow della DNA polimerasi I (Klenow fragment of DNA Polymerase I). Questo enzima aggiunge nucleotidi al primer di sequenziamento in modo complementare al filamento stampo, mentre i nucleotidi (dNTPs: adenina dATPαS, timina dTTP, citosina dCTP e guanina dGTP) sono dispensati uno alla volta in modo sequenziale.
Ogni volta che viene aggiunto un nucleotide, se questo è complementare alla sequenza stampo, vengono ripetute le seguenti reazioni:
- Il nucleotide viene incorporato nel filamento nascente dal frammento di Klenow e rilascia una molecola di pirofosfato (PPi);
- Il pirofosfato è utilizzato dall’enzima ATP sulfurilasi (ATP sulfurylase) per produrre ATP in modo equimolare;
- L’ATP è utilizzato dalla luciferasi (luciferase) per ossidare la luciferina ed emettere luce;
- L’apirasi (apyrase) rimuove i nuclueotidi non incorporati e l’ATP residuo.
Attenzione! dATP è sostituito con dATPαS (deoxyadenosine alfa-thio triphosphate) poiché può essere utilizzato dalla DNA polimerasi I, ma non è riconosciuta dalla luciferasi!

Metodo: preparare il DNA al pirosequenziamento
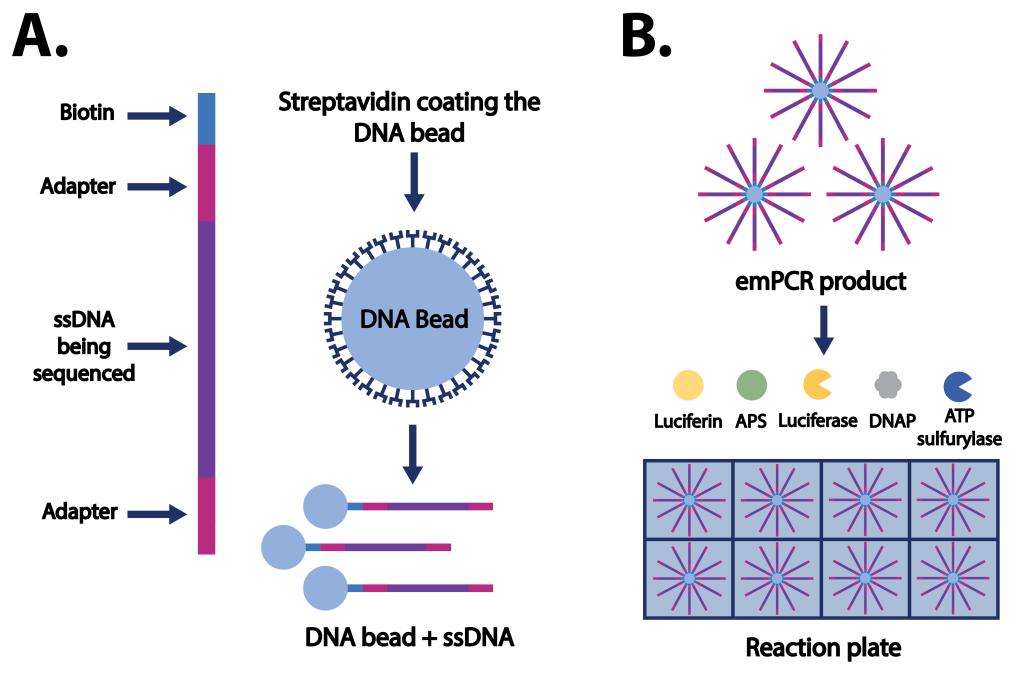
Prima del pirosequenziamento vero e proprio, il DNA da sequenziare viene frammentato e amplificato per creare una libreria di frammenti da sequenziare.
Il DNA è ridotto in frammenti di 300-800bp e alle estremità vengono ligati degli adattatori (con sequenza nota) di cui uno biotinilato. Questi frammenti di DNA a doppio filamento (dsDNA) vengono denaturati per ottenere DNA a singolo filamento (ssDNA) biotinilati ad una estremità. Questa estremità permette di immobilizzare il filamento a biglie ricoperte di streptavidina, grazie al legame avidina-biotina. In condizioni diluite, ogni biglia è lega un solo frammento di DNA a singolo filamento.
Ora ogni biglia ha una molecola d DNA da sequenziare, ma il segnale di una singola copia di DNA non è rilevabile dalla CCD (charge coupled device) camera. Quindi dobbiamo amplificare il filamento di DNA con la emulsion PCR (emPCR). La miscela di reazione è emulsionata per separare ogni singola biglia in una goccia di acqua in olio. In questo modo nel volume di reazione si creano molte gocce, e in ogni goccia ha luogo una PCR. Adesso le biglie sono ricoperte dalle molte copie della stessa sequenza legate covalentemente perché queste biglie sono anche ricoperte da primer per PCR.
Infine, denaturiamo per avere ssDNA con la stessa sequenza. Questi sono filamenti complementari al DNA da sequenziare cosicché il risultato del pirosequenziamento, ovvero i nucleotidi letti, è esattamente la sequenza d’interesse! Le biglie sono poi distribuite sulla piastra di sequenziamento. La piastra presenta delle celle in modo che ogni biglia occupi nello spazio un punto preciso e iniziano le reazioni del pirosequenziamento.
Risultati attesi: pirogramma
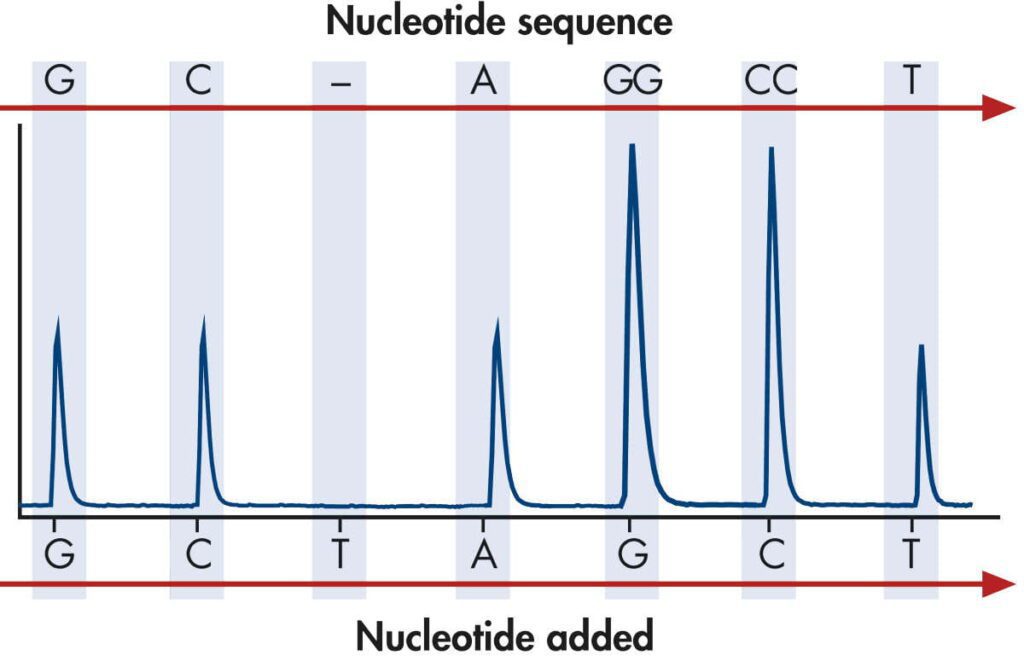
Durante il pirosequenziamento, la luce è registrata dalla CCD camera ed è convertita in un picco di segnale. L’intensità del segnale è proporzionale al numero di nucleotidi identici incorporati, perchè sono incorporati nello stesso step di sequenziamento. La sequenza di DNA può essere determinata dallo schema di luce che risulta dalla aggiunta dei nucleotidi in sequenza nella camera di reazione. In sintesi, si genera un pirogramma che un software specifico converte in sequenza nucleotidica. Il pirosequenziamento restituisce una sequenza di circa 100-400bp.
Limitazioni del pirosequenziamento
Il principale limite del pirosequenziamento è sequenziare regioni costituite da un solo tipo di nucleotide. Tutti e quattro i nucleotidi danno lo stesso segnale luminoso, e la luce emessa è proporzionale al numero di nucleotide aggiunti. Quando sono presenti due A consecutive, vengono incorporate due T e il segnale emesso è doppio rispetto al singolo nucleotide. Se sono presenti omopolimeri (regioni costituite da un solo tipo di nucleotide), si formano molte molecole di pirofosfato e molta luce che satura il segnale. Quindi vengono perse delle informazioni e la sequenza non è più corretta.
Quality Control
Come negli altri metodi di sequenziamento, sequenziare una sola volta un filamento può portare ad errori. Di conseguenza, serve un adeguato coverage (copertura, numero di volte che una singola posizione/nucleotide è identificato), per attribuire la giusta sequenza al filamento di DNA.
Fonti
- Ahmadian A, Ehn M, Hober S. Pyrosequencing: history, biochemistry and future. Clin Chim Acta. 2006 Jan;363(1-2):83-94. doi: 10.1016/j.cccn.2005.04.038. Epub 2005 Sep 13. PMID: 16165119.
- Harrington CT, Lin EI, Olson MT, Eshleman JR. Fundamentals of pyrosequencing. Arch Pathol Lab Med. 2013 Sep;137(9):1296-303. doi: 10.5858/arpa.2012-0463-RA. PMID: 23991743.
- https://www.qiagen.com/us/knowledge-and-support/knowledge-hub/technology-and-research/pyrosequencing-resource-center/pyrosequencing-technology-and-platform-overview
- https://apollo-institute.org/pyrosequencing/
Crediti immagini
- Immagine in evidenza: https://geneticsunzipped.com/transcripts/2021/9/23/future-of-DNA-sequencing
- Figura 1: https://meridian.allenpress.com/aplm/article/137/9/1296/193658/Fundamentals-of-Pyrosequencing
- Figura 2: https://apollo-institute.org/pyrosequencing/
- Figura 3: https://www.qiagen.com/us/knowledge-and-support/knowledge-hub/technology-and-research/pyrosequencing-resource-center/pyrosequencing-technology-and-platform-overview