Gli elementi genetici trasponibili sono frammenti di DNA che si possono spostare all’interno di una singola cellula da una posizione all’altra del cromosoma o dal cromosoma a un plasmide e viceversa.
Trasposizione
Questo processo di spostamento di DNA da un punto a un altro del genoma prende il nome di trasposizione e avviene con un meccanismo di ricombinazione sito-specifica in corrispondenza del sito di trasposizione. Questo sito contiene specifiche sequenze di DNA riconosciute da enzimi specializzati come le trasposasi o le ricombinasi sito-specifiche. I principali elementi genetici trasponibili sono le sequenze di inserzione, i trasposoni e gli integroni, gli elementi integrativi e coniugativi.
Le sequenze di inserzione (IS) contengono il gene per la trasposasi e, alle estremità, le sequenze invertite e ripetute (IR) lunghe poche decine di paia di basi, necessarie per lo spostamento.
Trasposoni
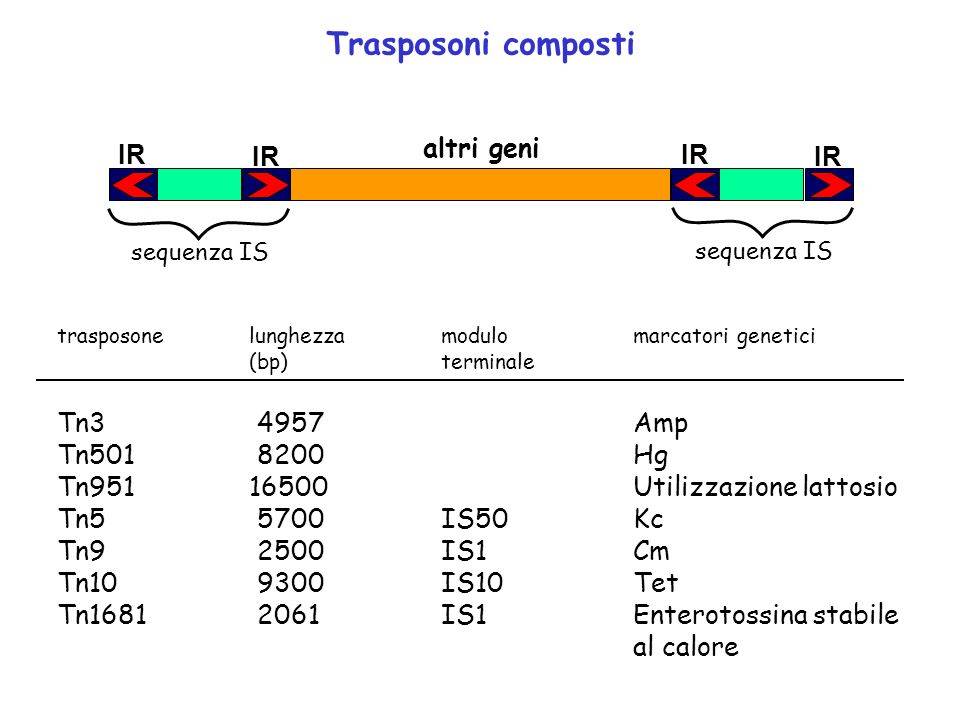
I trasposoni, in aggiunta alle IR e al gene per la trasposasi, contengono un numero variabile, a volte grande, di geni che conferiscono fenotipi particolari. Le IS sono degli elementi trasponibili semplici della lunghezza di circa 1000 bp capaci di inserirsi in punti particolari del genoma batterico.
Un genoma batterico può contenere varie copie di una stessa IS integrate in posizioni diverse: l’integrazione all’interno di un gene o di un operone (gruppo di geni la cui espressione è sotto il controllo di uno stesso promotore di trascrizione) può portare all’inattivazione del gene o dell’operone stesso. Tra i geni veicolati dai trasposoni ricordiamo quelli che conferiscono resistenza antibiotica, quelli che producono tossine, quelli che codificano fattori di virulenza. Il trasposone più semplice è quello costituito da un gene o da un gruppo di geni fiancheggiati da due identiche IS, la cui trasposasi catalizza lo spostamento dell’intero elemento. Un esempio è rappresentato dal trasposone Tn5, che contiene geni di resistenza antibiotica fiancheggiati da due elementi IS.
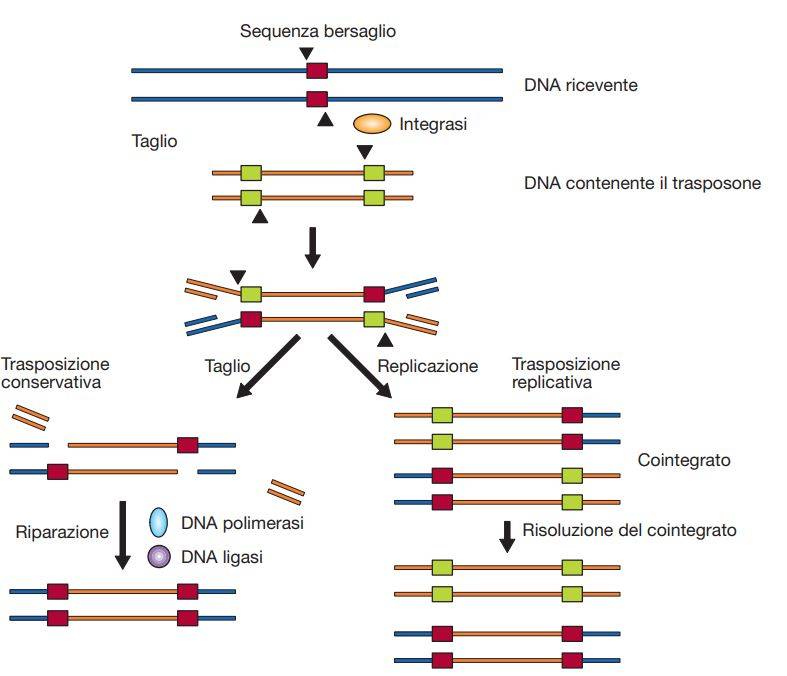
Un gruppo differente di trasposoni come quelli coniugativi non presenta elementi IS all’estremità ma solo delle sequenze invertite ripetute riconosciute dalle ricombinasi sito-specifiche che catalizzano la trasposizione. Spesso i trasposoni possono diventare di grandi dimensioni grazie alla fusione di più trasposoni tra loro e questo può portare alla nascita di elementi genetici trasponibili codificanti tanti e diversi determinanti di resistenza antibiotica. Durante il processo di trasposizione, la trasposasi riconosce la sequenza del sito bersaglio e produce un taglio a singolo filamento; il trasposone subisce anch’esso dei tagli e si inserisce nella sequenza bersaglio dove il DNA viene risaldato con conseguente duplicazione della sequenza bersaglio.
Nella trasposizione conservativa il trasposone viene perso dalla sede originaria e acquisito in una nuova sede, mentre nella trasposizione replicativa una copia rimane nel sito originario e una va nel nuovo sito. Entrambi i tipi di trasposizione prevedono i tagli a singolo filamento e integrazione del trasposone. Nella trasposizione conservativa il sito bersaglio viene tagliato di nuovo e le interruzioni nei filamenti vengono riparate con formazione di una ripetizione diretta che fiancheggia il trasposone. Nella trasposizione replicativa il processo di riparazione inizia quando il trasposone è legato sia al sito originario che a quello bersaglio e ciò porta alla formazione di una struttura detta cointegrato, che viene poi risolta con il rilascio del trasposone originario e la presenza di una nuova copia del trasposone nel sito bersaglio.
Integroni
Gli integroni sono elementi genetici trasponibili che si inseriscono nel cromosoma batterico e spesso anche in plasmidi o in trasposoni e pertanto sono facilmente mobilizzabili tra specie batteriche diverse. Questi elementi sono costituiti da un gene per l’integrasi e una sequenza specifica di attacco, entrambi coinvolti nel processo di ricombinazione sito-specifica. La particolarità degli integroni è la presenza di una sequenza specifica di DNA che permette l’acquisizione di nuovi geni, detti cassette, da fonti diverse e di esprimerli sotto il controllo di un promotore residente.
Spesso i nuovi geni che si inseriscono sono determinanti di resistenza antibiotica o di virulenza e il loro numero può essere grande. Gli integroni sono diffusi in parecchie specie batteriche tra cui Pseudomonas aeruginosa e possono facilmente essere trasferiti in batteri patogeni per scambio genico orizzontale e selezionati dall’uso di antibiotici nella pratica clinica.
Elementi integrativi e coniugativi
Gli elementi integrativi e coniugativi sono elementi genetici mobili capaci di auto-trasferirsi e hanno caratteristiche in comune con i plasmidi, con cui condividono il trasferimento tramite coniugazione, e con i batteriofagi, con i quali condividono la capacità di integrarsi in punti specifici del cromosoma della cellula ospite. La struttura degli ICE contiene un’impalcatura composta da tre moduli i cui geni assicurano il mantenimento, la disseminazione e la regolazione. Questi moduli possono essere scambiati tra gli elementi genetici mobili e tra questi ultimi e il cromosoma della cellula ospite.
Come i batteriofagi temperati, gli ICE si integrano in un replicone dell’ospite, il cromosoma o un plasmide, il quale assicura il trasferimento verticale alla progenie batterica. Il modulo di mantenimento (o integrazione) contiene un gene che codifica l’integrasi (Int) che promuove l’integrazione dell’ICE nel cromosoma (o nel plasmide). L’integrasi appartiene alla famiglia delle ricombinasi ed è un enzima che catalizza la ricombinazione tra una sequenza specifica attP (phage attachment site), presente nel modulo di integrazione dell’ICE, e una sequenza bersaglio, identica o altamente omologa, attB (bacterial attachment site), presente nel cromosoma dell’ospite.
Le integrasi degli ICE più comuni sono tirosine ricombinasi e sono simili alla integrasi del fago lambda (λ) di Escherichia coli. Una forma circolare dell’ICE si integra nel cromosoma mediante ricombinazione tra le sequenze attP e attB e genera due sequenze di giunzione ICE-cromosoma, attachment left (attL) e attachment right (attR). Nel modulo di integrazione spesso è presente un gene codificante per l’escissionasi (Xis) che è richiesta per l’escissione dell’ICE dal cromosoma. L’escissionasi facilita la ricombinazione, catalizzata dall’integrasi, tra le sequenze di giunzione attL e attR.
Alcuni ICE hanno degli attB variabili: ad esempio gli ICE Tn5251 di S. pneumoniae e Tn916 di Enterococcus faecalis si integrano in più punti del cromosoma a livello di sequenze ricche di nucleotidi A e T. Altri ICE, come il Tn5253 di S. pneumoniae e l’SXT di Vibrio cholerae, si integrano in un unico punto specifico del cromosoma, il Tn5253 nella porzione 5′ del gene essenziale rbgA e l’SXT nella porzione 5′ del gene prfC. L’inserzione del Tn5253 in rbgA non compromette la vitalità del ceppo batterico che lo ospita in quanto la duplicazione del sito bersaglio, conseguente alla integrazione dell’ICE, ristabilisce la coding sequence del gene.
Va detto che in assenza degli attB specifici di integrazione, sia il Tn5253 che l’SXT si integrano altrove nel cromosoma batterico. ICE simili possono contenere moduli di mantenimento differenti, ad esempio il Tn916 e il Tn5397 di Clostridium difficile (entrambi veicolanti il gene tet(M) che conferisce resistenza alla tetraciclina) hanno dei moduli di regolazione e disseminazione con un’identità a livello di sequenza nucleotidica superiore all’80%, ma moduli di integrazione differenti. Tn916 codifica una tirosina ricombinasi (Int) e un’escissionasi (Xis), mentre Tn5397 codifica un’unica grande serina ricombinasi (TndX) che catalizza l’inserzione dell’elemento genetico in un sito specifico del cromosoma contenente al centro un dinucleotide GA.
Il modulo di disseminazione (o coniugazione) contiene i geni che codificano le proteine coinvolte nel processo di coniugazione durante il quale, in seguito a contatto cellulare, il DNA dell’elemento genetico mobile viene trasferito dalla cellula donatrice a quella ricevente. Verosimilmente la maggior parte degli ICE trasferisce un singolo filamento di DNA, ad eccezione di alcuni che trasferiscono il doppio filamento di DNA. Anche i moduli di disseminazione sono diversi tra i vari ICE ed elementi diversi possono avere moduli simili, ad esempio i geni della coniugazione dell’SXT sono simili a quelli del plasmide coniugativo F di E. coli. Il sistema di trasferimento di parecchi ICE può essere parassitato dagli elementi mobilizzabili, che sono elementi non in grado di trasferirsi autonomamente ma che possono utilizzare gli apparati di trasferimento degli ICE. In questi casi i moduli di disseminazione dell’elemento mobilizzabile e dell’ICE che lo mobilizza contengono un’origine di trasferimento (oriT) differente.
Alcuni elementi incapaci di trasferirsi per coniugazione da cellula a cellula si sono integrati, nel corso dell’evoluzione, all’interno di altri elementi che ne permettono il trasferimento. L’elemento genetico Ωcat(pC194) di S. pneumoniae fa parte dell’ICE composito Tn5253 che ne assicura il trasferimento a diverse specie batteriche. L’Ωcat(pC194), che veicola il gene cat di resistenza al cloramfenicolo, autonomamente è capace solo di mobilità intracellulare. L’elemento si escinde dal Tn5253, produce un intermedio circolare e si integra per ricombinazione omologa altrove nel cromosoma di S. pneumoniae. Vale la pena ricordare che il Tn5251, che veicola il gene tet(M), è un ICE che fa parte anch’esso dell’elemento composito Tn5253, ma oltre ad essere trasferito da quest’ultimo è capace di trasferimento coniugativo autonomo a specie batteriche diverse. Il modulo di regolazione contiene i geni che regolano il trasferimento degli ICE. Il trasferimento del Tn916 e dell’ICE CTnDOT di Bacteroides, che conferiscono resistenza alla tetraciclina, è indotto da concentrazioni subinibitorie di tetraciclina, ma il meccanismo di induzione è differente.
La tetraciclina innesca la disseminazione di ICE che conferiscono alle nuove cellule ospiti la capacità di sopravvivere in presenza di concentrazioni dell’antibiotico che risultavano letali prima dell’acquisizione dell’elemento genetico. L’ICE SXT codifica la proteina SetR, che è un ortologo del repressore CI del fago λ che controlla la lisogenia (integrazione del DNA fagico in quello cromosomale).
Verosimilmente, come per il fago λ, l’induzione della risposta SOS (sistema di riparazione del DNA danneggiato) da parte della mitomicina C (chemioterapico che induce danni nel DNA) riduce la repressione operata da SetR e induce il trasferimento di SXT di circa 400 volte. Nell’ICESt1 di Streptococcus thermophilus l’espressione di un repressore inattivato dalla risposta SOS ne regola il trasferimento. La risposta batterica globale al danneggiamento del DNA si traduce nella regolazione del trasferimento di vari ICE. L’impalcatura dell’ICE con i suoi 3 moduli funzionali può acquisire geni codificanti funzioni diverse, che possono così essere disseminati nella popolazione batterica. Tra questi ricordiamo quelli che conferiscono resistenza antibiotica e quelli coinvolti nella virulenza. Alla stessa stregua dei plasmidi coniugativi, gli ICE sono molto importanti da un punto di vista clinico, perché responsabili della diffusione di questi geni tra batteri patogeni anche non correlati.
Fonti
- Didelot, X. et al. (2016), «Within-host evolution of bacterial pathogens», Nat Rev Microbiol, 14(3), pp. 150-62.
- Fleischmann, R.D. et al. (1995), «Whole-genome random sequencing and assembly of Haemophilus influenzae Rd», Science, 269(5223), pp. 496-512.
- Goodwin, S. et al. (2016), «Coming of age: ten years of next-generation sequencing technologies», Nat Rev Genet, 17(6), pp. 333-351.
- Johnson, C.M. et al. (2015), «Integrative and Conjugative Elements (ICEs): what they do and how they work», Annu Rev Genet, 49, pp. 577-601.
- Margulies, M. et al. (2005), «Genome sequencing in microfabricated high-density picolitre reactors», Nature, 437(7057), pp. 376-380.
- Ronaghi, M. et al. (1996), «Real-time DNA sequencing using detection of pyrophosphate release», Anal. Biochem., 242(1), pp. 84-89.
- Sanger, F. et al. (1977), «DNA sequencing with chain-terminating inhibitors», Proc Natl Acad Sci USA, 74(12), pp. 5463-5467.
- Sboner, A. et al. (2011), «The real cost of sequencing: higher than you think!» Genome Biology, 12:125.
- Service, R.F. (2006), «Gene sequencing. The race for the $1000 genome», Science, 311(5767),pp. 1544-1546.