Definizione
Prima di introdurre il concetto di metabolomica è opportuno definire il concetto di metaboloma, ossia la collezione univoca dell’insieme di metaboliti (molecole a basso peso molecolare) prodotti durante un determinato metabolismo a livello cellulare, tissutale o sistemico.
La metabolomica, pertanto, è la disciplina scientifica che si occupa di identificare gli specifici metabolomi che descrivono i singoli processi biologici.
Nel complesso delle scienze -omiche, pertanto, la metabolomica si posiziona alla base di una piramide al cui vertice troviamo l’analisi dell’informazione genica, la genomica, per poi seguire con la trascrittomica o analisi dell’espressione genica attraverso la collezione di mRNAs. Scendendo nella piramide, passiamo all’analisi di funzionalità genica con la proteomica che studia l’insieme delle proteine tradotte da specifiche sequenze geniche.
La metabolomica completa l’analisi verticale di un determinato processo biologico identificando i prodotti chimici, i metaboliti, a valle di un determinato quadro genetico.
Cenni storici
L’idea che ogni individuo sia caratterizzato da un cosiddetto profilo metabolico, in inglese metabolic fingerprint, è stata introdotta per la prima volta da Roger Williams nel lontano 1940, il quale ipotizzò l’esistenza di una firma metabolica nella schizofrenia, a seguito dell’analisi cromatografica di campioni di saliva e urina.
Tuttavia, fu solo nel 1971 quando si è introdotto il concetto di profilo metabolico, grazie ai primi studi effettuati dagli scienziati Horning, Linus Pauling e Arthur B. Robinson, che condussero allo sviluppo di metodi di analitici di GC-MS per il monitoraggio dei metaboliti nell’urina.
Contemporaneamente, nel 1971, Seeley e collaboratori dimostrarono l’utilizzo della tecnica NMR (risonanza magnetica nucleare) per l’identificazione dei metaboliti presenti in campioni biologici non trattati. L’aumento della sensibilità di questa tecnica la rende ad oggi una delle più utilizzate per l’identificazione di una firma metabolomica in campioni biologici di varia natura.
Nel 1995 avvenne l’incorporazione della LC/MS (liquido cromatografia associata a spettrometria di massa) tra le tecniche utilizzate negli studi di metabolomica grazie al contributo dei chimici Gary Siuzdak, Richard Lerner e Benjamin Cravatt, i cui esperimenti portarono all’identificazione dell’oleamide, un metabolito dalle funzioni rilevanti nell’induzione del sonno.
Nel 2005, è stato quindi inaugurato il primo database dei dati di metaboliti umani identificati tramite spettrometria di massa, il METLIN, sviluppato dai ricercatori del laboratorio di Siuzdak che attualmente contiene più di 450000 metaboliti.
Il Progetto del Metaboloma Umano (HMP)
A gennaio del 2007, il progetto Human Metabolome Project (HMP) completò il primo draft del metaboloma umano, attraverso la realizzazione dello Human Metabolome Database (HMDB), che incorpora al suo interno 4 database addizionali, DrugBank, T3DB, SMPDB and FooDB.
Lo HMDB contiene in tutto circa 11422 metaboliti, dei quali DrugBank contiene informazioni equivalenti a circa 2280 farmaci e metaboliti di farmaci, T3DB contiene informazioni riguardanti 3670 tossine comuni e inquinanti ambientali, SMPDB contiene diagrammi per 25.000 vie metaboliche e malattie umane, mentre FooDB contiene informazioni equivalenti su 28.000 componenti alimentari e additivi alimentari .
Tecnologie analitiche
Quando parliamo di metabolomica, in realtà possiamo fare riferimento a due tipologie analitiche ben distinte: il metodo quantitativo e il metodo chemiometrico.
Il metodo quantitativo o “targeted detection”, che è stato il primo a inserirsi nella scena, è basato sull’identificazione e quantificazione univoca di determinati metaboliti, per i quali esiste uno standard di riferimento.
Al contrario, il metodo chemiometrico o “untargeted detection”, utilizza le stesse tecnologie analitiche precedenti, ma si basa sull’identificazione di un profilo di metaboliti tramite l’applicazione di metodi statistici di predizione.
Per farla breve, è come se pensassimo di andare a pescare con una sola canna da pesca alla ricerca di una succulenta trota o se invece pensassimo di gettare delle reti capienti nel nostro laghetto. Nel primo caso torneremo a casa con la nostra trota, nel secondo con una svariata quantità di pesci, tra cui probabilmente la nostra trota.
Evidentemente, il secondo metodo è molto più speculativo e ingegnoso per quanto riguarda l’analisi statistica dei dati ottenuti. Si richiede infatti lo sviluppo di tecniche sempre più avanzate di analisi multivariata che consentano, davanti ad uno spettro cromatografico di innumerevoli picchi, di applicare delle teorie predittive per etichettarli e quantificarli (Immagine 1).

Tecniche di analisi multivariata
Una delle principali tecniche di analisi multivariata è la PCA (Principal Component Analysis) che consente di identificare le componenti principali di un sistema complesso, ossia l’insieme dei metaboliti che discriminano campioni biologici differenti.
Una variazione di questa tecnica di analisi qualitativa è ad esempio la OPLS-DA che, a differenza della PCA, è in grado di quantificare la variabilità che esiste all’interno di ciascun campione.
Ovviamente a partire da questi due approcci esiste un’ampia gamma di possibilità analitiche che permette di identificare una firma multi-variabile, alias una firma di metaboliti tra classi biologiche differenti.
Metabolomica per il controllo qualità
Uno dei campi di applicazione della metabolomica è il controllo qualità sensoriale di bevande e alimenti. La valutazione della qualità sensoriale di un determinato alimento o bevanda ha per molti anni limitato quest’area analitica alla valutazione soggettiva. L’applicazione della metabolomica in questo settore ha invece permesso l’identificazione di una serie di composti chimici che definiscono in maniera univoca e oggettiva, il buon gusto di alimenti o bevande.
Un esempio proviene dallo screening di autenticazione dei composti significativi nel caffè più costoso del mondo: quello derivante dalla defecazione della civetta di palma, il caffè Kopi Luwak.

Grazie ad un’analisi metabolomica discriminante tra chicchi di caffè Kopi Luwak e chicchi di caffè normale (Immagine 2), gli scienziati hanno ottenuto una firma metabolica standard applicabile nella fase di autenticazione di questo costoso caffè: un mix di acido malico, acido glicolico e acido citrico.
Metabolomica per la prevenzione e predizione di patologie
Tuttavia, il campo in cui la metabolomica ha riscontrato maggior successo e utilità è quello della prevenzione e prognosi di patologie multifattoriali, in cui è necessaria la descrizione metabolica di un fenotipo specifico.
A differenza delle malattie genetiche ereditarie, per cui la disponibilità di una quantità limitata di biomarker potrebbe essere sufficiente per la diagnosi e prognosi della patologia, nelle malattie multifattoriali, come l’obesità, si aspira all’identificazione di una firma metabolica, costituita da centinaia di metaboliti (Immagine 3).
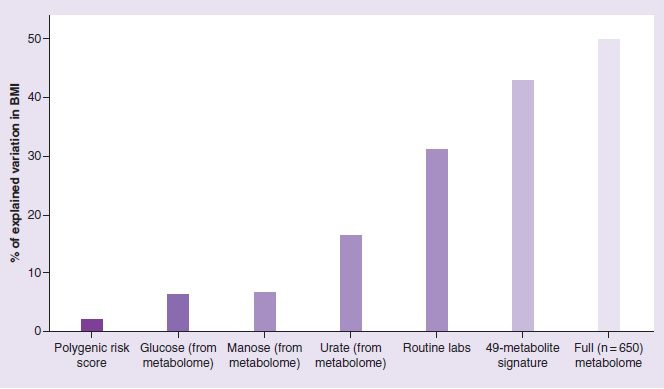
In questo contesto, la disponibilità dell’HMDB risulta di vitale importanza per la comprensione del ruolo svolto da ciascun metabolito all’interno dei processi biologici a capo di patologie così complesse come l’obesità
Nutrimetabolomica: il futuro della medicina personalizzata
In conclusione e in vista del nuovo approccio personalizzato della medicina, oggi siamo in grado di parlare anche di nutrimetabolomica. Il concetto base consiste nella possibilità di identificare, a livello personalizzato, la nostra mappa metabolica. L’obbiettivo? Quello di comprendere, al dettaglio del singolo metabolito, il proprio metabolismo, in modo tale da stabilire un’alimentazione appropriata per le esigenze di ciascun fenotipo.
Un esempio: l’analisi del nostro profilo metabolico ci allerta riguardo una particolare predisposizione all’insorgenza di sindrome metabolica. Ebbene, sapendo questo e agendo in anticipo posso curare la mia dieta e il mio stile di vita in maniera personalizzata al fine di ingannare il nostro ingranaggio metabolico.
Una visione troppo futurista? Benvenuti nel mondo delle –omiche e della loro potenzialità.
Fonti
- Daviss B (April 2005). Growing pains for metabolomics. The Scientist. 19 (8): 25–28.
- Hollywood K, Brison DR, Goodacre R (September 2006). “Metabolomics: current technologies and future trends”. Proteomics. 6 (17): 4716–23.
- Holmes E, Antti H (December 2002). “Chemometric contributions to the evolution of metabonomics: mathematical solutions to characterising and interpreting complex biological NMR spectra”. The Analyst. 127 (12): 1549–57.
- Cirulli ET, Guo L, Leon Swisher C et al. Profound perturbation of the metabolome in obesity associates with health risk. Cell Metab. doi:10.1016/j.cmet.2018.09.022 (2018)
- Marcone MF. Composition and properties of Indonesian palm civet coffee (Kopi Luwak) and Ethiopian civet coffee. Food Research International (Ottawa, Ont.). 2004 ;37(9):901-912. DOI: 10.1016/j.foodres.2004.05.008.