Dopo la scoperta del meccanismo di RNAinterference (RNAi) da parte di Fire e Mello nel 1998, l’interesse per una sua applicazione nella difesa delle piante è cresciuto molto. Non tutti hanno ben presente i meccanismi che si celano dietro questa tecnica, o i suoi vantaggi e svantaggi. Scopriamo insieme come funziona questo meccanismo e come può essere utilizzato.
I sRNAs
Prima di parlare di RNAi è giusto chiarire cosa sono e cosa fanno i sRNA nelle cellule.
Gli smallRNA, o sRNA, sono un gruppo di RNA con dimensioni molto ridotte (circa 20-24 nucleotidi), presenti all’interno delle cellule, dove svolgono funzioni molto importanti. Il termine sRNA tuttavia comprende al suo interno una ampia varietà di molecole che si distinguono tra loro per il regno e la classe di organismi in cui sono presenti e le attività che svolgono all’interno del metabolismo cellulare.
Tra i sRNAs ricordiamo:
- microRNAs o miRNAs: sono importanti principalmente nei mammiferi nel ruolo di silenziamento genico
- piwiRNAs o piRNAs: si originano da precurosi a single strand, sono importanti nello sviluppo delle cellule germinali grazie alla proteina piwi, che chiameremo anche AGO.
- longer non-codingRNAs o ncRNAs: di recente scoperta la loro fuzione resta ancora sconosciuta
- small nuclear RNAs o snRNAs: regolano la trascrizione a livello di splicing
- snoRNAs o small nucleolar RNAs: partecipano ai fenomeni di maturazione dell’RNA
- small cytoplasmatic RNAs o scRNAs: sono coinvolti nel sorting proteico
- siRNAs small interferingRNAs: sono sempre piccoli RNA come i miRNA, ma a differenza di questi sono di origine esogena, in genere virale. L’altra fondamentale differenza con i miRNA è la specificità d’azione che richiede una complementarietà tra i filamenti perfetta.
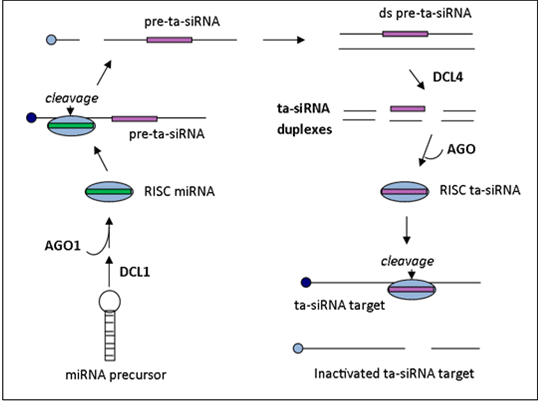
Cos’è l’RNAi?
L’RNAi è un meccanismo naturalmente presente in tutte le cellule eucariotiche utilizzato per il silenziamento di alcuni geni. Solitamente i geni che sono controllati da questo meccanismo sono geni virali e quindi di derivazione extracellulare o sequenze trasponibili. Per questa ragione questo tale viene anche considerato come “l’arma di difesa” di molti eucarioti nei confronti dei virus.
Il meccanismo può essere classificato come un regolatore post-trascrizionale dell’espressione genica dell’individuo. In particolare effettua un silenziamento delle sequenze trascritte definito Post-Transcriptional Gene silencing (PTGS).
L’aspetto che ha portato allo sviluppo delle biotecnologie nella difesa delle colture di questo meccanismo è l’identificazione, in alcune piante, dell’utilizzo dei siRNAs come effettori. Tali effettori vengono rilasciati dalla pianta per bloccare il patogeno, in questo caso mediante il silenziamento di alcuni geni nel microrganismo. I geni silenziati sono fondamentali o per la vita o per l’attività infettiva del patogeno, e viene quindi arrestato il decorso della malattia.
Il meccanismo appena descritto è noto comunemente come Host Induced Gene Silencing (HIGS).
Cosa comporta l’RNAi nella pratica
Le prime applicazioni di questo metodo si sono avute con piante transgeniche. Queste piante sono in grado di produrre dei dsRNA specifici per un patogeno chiave, in grado di proteggerle dal suo attacco.
L’utilizzo dei dsRNA (double strand RNA, Fig. 2) è risultato fondamentale, in quanto i siRNA sono molecole facilmente degradabili dalle endonucleasi dei patogeni. La pianta stessa infatti naturalmente non produce direttamente siRNA ma, come vedremo, dsRNA che ne siano precursori e che si modifichino in siRNA solo nel patogeno.
Vista la difficolta del processo di trans-genesi si sono testati altri metodi per valutare l’efficacia dei dsRNA nei confronti dei patogeni. In particolare si sono applicati mediante trattamento spray sulla superfice fogliare o inoculati mediante una siringa all’interno dei piccioli fogliari. In entrambi i casi i risultati sono stati positivi. Tuttavia, l’applicazione spray, nonostante la ridotta persistenza sulla foglia, sembra essere il metodo che fornisce i maggiori risultati in proporzione ai costi di distribuzione, deriva da questo il termine di Spray Induced Gene Slicencing (SIGS).
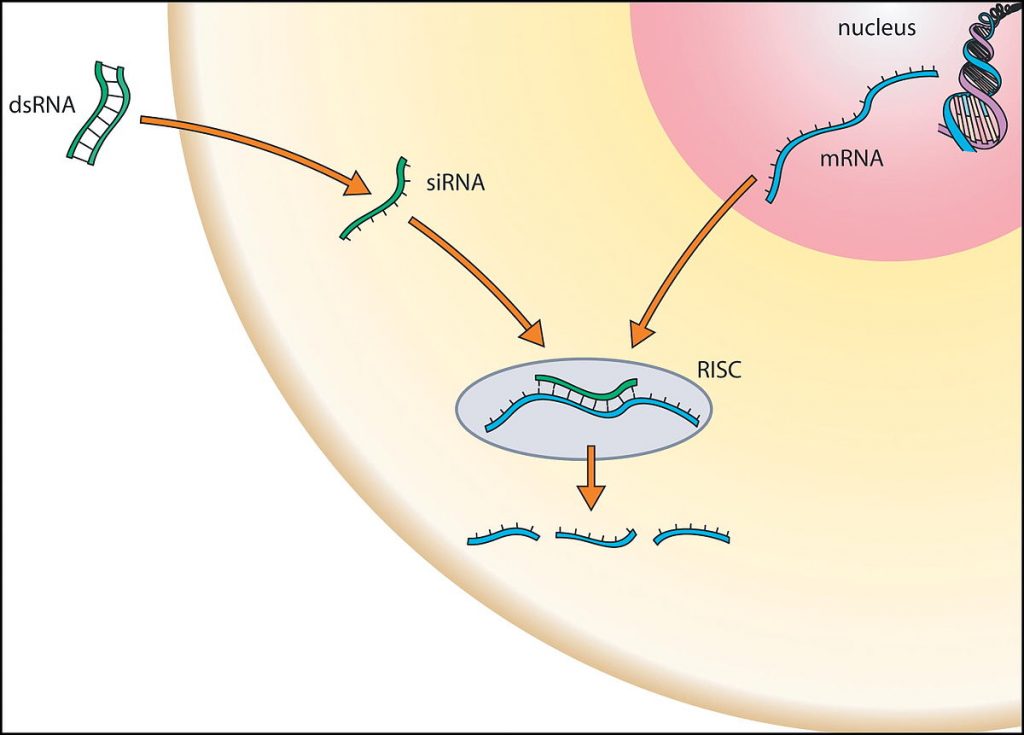
Meccanismo RNAi
Il meccanismo dell’RNAi (Fig. 3) ha origine dalla presenza di dsRNA all’interno della cellula. Ricordiamo che questi ultimi possono essere trascritti perché patrimonio del genoma dell’individuo o di derivazione esogena.
Per chiarezza di interesse descriveremo il meccanismo di RNAi dei funghi.
All’interno del citoplasma la presenza dei dsRNA viene riconosciuta da parte di alcuni ligandi, DCL1 e DCL2. Non appena avviene il riconoscimento, i DCL reclutano il complesso SAGA. Il complesso SAGA presenta un’attività istone acetiltransferasi che incrementa la trascrizione dei promotori DCL e AGO e attiva la macchina RNAi.
Per rompere il doppio filamento, il 5’ del dsRNA si ancora al dominio PAZ (PiwiArgonaute-Zwille/Pinhead) e alle DCL. Grazie a questo legame le successive RNAasiIII intervengono, rompendo anche i legami Ribosio-PO42- e portando a siRNA di 21-25 nucleotidi con al 5’ un monofosfato e al 3’ due nucleotidi sporgenti.
Appena il siRNA viene prodotto, assieme al complesso AGO recluta QIP (QDE-e-INTERACTING PROTEIN). Questo porta alla formazione del complesso di silenziamento indotto dall’RNA (RISC) RNA-Induced Silencing Complex.
QIP è in grado di riconoscere e degradare la sequenza legata al siRNA con la sua attività esonucleasica. Il complesso RISC una volta attivato cerca trascritti che siano complementari con i siRNA silenziando la sequenza guida.
Quando un mRNA si appaia con la sequenza guida del RISC, l’attività esonucleasica viene attivata.
RdRP e hpRNA
In alcuni microrganismi, durante la lunga elaborazione, i siRNAs possono fungere da primer e attivare la RNA-dependent RNA-Polimerasys (RdRP). I siRNAs, appaiandosi con mRNA complementari, agiscono da primer per la sintesi del secondo filamento mediante proprio il reclutamento degli RdRP.
Il secondo dsRNA prodotto dall‘RdRP richiama un’altra attività DCL e un nuova formazione del complesso RISC, portando all’amplificazione del silenziamento mediato dal meccanismo RNAi.
Nelle piante ingegnerizzate, le molecole che vengono prodotte non sono propriamente dsRNA, nonostante svolgano la stessa funzione. Quelli che vengono definiti hairpinRNA (hpRNA) sono in grado comunque di attivare il meccanismo di RNAi anche se mediante dei siRNA di soli 21 nucleotidi.
Il trasporto degli hpRNA dal nucleo al citoplasma dove possono svolgere la loro funzione è mediato da proteine che prendono il nome di HASTY.
Come viene trasferito il dsRNA al patogeno
Nonostante l’origine e il meccanismo di funzionamento dell’RNAi siano abbastanza chiari, il trasferimento dei dsRNA da pianta a patogeno rimane un’incognita.
Negli animali, dove questo fenomeno è stato maggiormente approfondito, sembra che questa assimilazione sia una endocitosi mediata da esosomi. Alcune vescicole di membrana contenenti sRNA si fondono con la membrana plasmatica fino al trasferimento nel citoplasma nel patogeno.
In alternativa i sRNA possono coinvolgere trasportatori transmemebrana senza l’utilizzo delle vescicole derivanti dall’ospite. I sRNAs riscontrati nello spazio extracellulare tendono ad associarsi con le lipoproteine ad alta densità e trasferirsi nelle cellule riceventi.
All’interno delle piante invece, si è visto che i sRNA si muovo sia tra le varie cellule mediante i plasmodesmi sia nell’intero individuo passando attraverso i vasi.
I processi che vedono il trasferimento dei sRNA al patogeno sono molto selettivi, e per questo sono sicuramente esclusi metodi passivi come la diffusione.
Uno dei metodi ipotizzati è quello del trasferimento mediante vescicole extracellulari. Questa ipotesi è confutata dal fatto che sRNA e dsRNA, per quanto siano resistenti, difficilmente riescono a superare indenni le endonucleasi fungine.
Partendo da questo, sono stati identificati in Arabidopsis dei geni TETlike (Tetrasprina), TET8 e TET9, indotti dall’attacco di botrite. Una molecola che è stata colocalizzata con TET8 è una guanosina trifosfatasi derivante dal gene ARA6. Questa molecola è una GTPasi classficata nelle Rab5-like che rappresenta un Marcatore di corpi multivesciolari. Questo suggerisce che le vescicole extracellulari probabilmente derivano da corpi multivesciolari. Inoltre, le vesciole derivanti da TET8 possono essere considerate esosomi delle piante.
Il cerchio si chiude con l’abilità dimostrata nei funghi di assorbire gli esosomi delle piante. Per prova, alcuni sRNA delle piante trasportati mediante esosomi sono stati trovati nel citoplasma fungino.
Applicazione della tecnica
A livello di ricerca sono state molte le applicazioni dell’RNAi, meccanismo la cui efficacia è stata dimostrata nei confronti di virus, funghi e insetti.
Per quanti riguarda il mondo virale, interessante è stato l’utilizzo nei confronti del virus del mosaico dei cocomeri e del Bean Golden Mosaic Virus utilizzando come target il gene AC1. In Brasile l’utilizzo dell’RNAi nei confronti Pheseolus vulgaris è stato approvato anche in campo.
Molti sono stati gli approcci nei confronti di insetti come Leptinotarsa decemlineata e Diabrotica virgifera virgifera. In U.S.A. e in Brasile è stato approvato l’uso dell’HIGH nei confronti del gene DvSnf4. Questo gene risulta fondamentale nella degradazione dei lisosomi delle radici da parte di Diabrotica, infatti il suo silenziamento porta a malfunzionamenti gastrici dell’insetto.
Nel caso dei funghi, il meccanismo RNAi è stato provato nei confronti della peronospora della patata (Phytophthora infestans) e di alcune specie di Verticillum e Fusarium. L’efficacia maggiore tuttavia sembra essere stata data dal silenziamento del gene SSCHS codificante per una chitina sintasi in Slerotinia sclerotium.
Il grosso limite tecnico, nel caso dei SIGS, resta il ridotto periodo di copertura. Alcuni studi hanno infatti dimostrato un aumento della persistenza sulla foglia dei dsRNA se accoppiate con nanoparticelle di argilla.
Questa tecnica è stata testata anche per produrre piante in grado di sintetizzare specifici dsRNA con lo scopo di incrementare l’impollinazione incrociata, stimolando l’attività di alcuni impollinatori. Tra questi gli invertrebrati Apis mellifera, Eisenia adrei, Coleomegilla maculata e i vertebrati Gallus domesticus e Ictalurus punctatus.
Da sottolineare che le larve delle api che sono state sottoposte a dosaggi 10 volte superiori rispetto a quelli a cui sarebbero sottoposti in ambiente non hanno mostrato alcun aspetto negativo sulla crescita.
Nemmeno l’applicazione del dsRNA esogeno mediante metodo spray ha portato ad effetti negativi nei confronti delle api.
Vantaggi e svantaggi
La specificità delle sequenze dei meccanismi di RNAi porta a vantaggi impareggiabili. Sicuramente è una tecnologia che offre sicurezza e sostenibilità ambientale ponendosi come alternativa ai più comuni agrofarmaci.
I fattori limitanti di questo approccio sono sicuramente la difficoltà e la complessità dei protocolli di trasformazione, e le restrizioni legislative sulle piante ingegnerizzate. Come abbiamo già detto si potrebbe ovviare a questo limite attraverso la tecnica SIGS, tuttavia la legislazione europea in materia in modo molto grossolano la accomuna all’utilizzo di OGM.
Un altro grande vantaggio del meccanismo di RNAi è la stabilità del dsRNA nella fillosfera. Stabilità che si manifesta come visto anche in una maggior resistenza alle endonucleasi rispetto ai ssRNa.
Grandissimo vantaggio dei dsRNA è inoltre la ridotta persistenza di queste molecole nella pedosfera: in genere il tempo di degradazione al suolo è di massimo 24 ore. La degradazione si allunga a 96 ore nel caso di sistema di acqua naturale completo. Le nucleasi batteriche, in particolare RNasiIII sono principalmente responsabili della degradazione dei dsRNA nel suolo e nell’acqua.
L’utilizzo di questa tecnologia non crea assolutamente rischi ai nostri cibi; infatti tutti i nostri alimenti contengono già elevate quantità di sRNAs, come siRNAs, miRNAs, piRNAs e dsRNAs endogeni, prodotti da una varietà di fonti come virus naturalmente presenti nella pianta e trasposoni presenti nel genoma.
La maggior parte dei trattamenti attuali sugli alimenti come cottura, microronde, estrazione con solventi, trattamenti termici, fermentazione, acidificazione e alcalinizzazione, risultano in una degradazione del materiale genetico presente nelle cellule. Qualsiasi dsRNA o meccanismo di RNAi venga utilizzato non resiste ai classici trattamenti degli alimenti.
Questo aprirebbe anche la possibilità di utilizzarli come trattamenti post raccolta e nella conservazione degli alimenti.
Fonti
- Cai, Q., Qiao, L., Wang, M., He, B., Lin, F.-M., Palmquist, J., Huang, S.-D., Jin, H., 2018. Plants send small RNAs in extracellular vesicles to fungal pathogen to silence virulence genes. Science 360, 1126–1129.
- Mcloughlin, A.G., Walker, P.L., Wytinck, N., Sullivan, D.S., Whyard, S., Belmonte, M.F., 2018. Developing new RNA interference technologies to control fungal pathogens. Can. J. Plant Pathol. 40, 325–335.
- Nerva, L., Sandrini, M., Gambino, G., Chitarra, W., 2020. Double-stranded RNAs (dsRNAs) as a sustainable tool against gray mold (Botrytis cinerea) in grapevine: Effectiveness of different application methods in an open-air environment. Biomolecules 10, 200.
Fonti immagini
- Immagine in evidenza: httpscommons.wikimedia.orgwikiFileRNAi_overview.png
- Figura 1: httpscommons.wikimedia.orgwikiFileTa-siRNA_Biogenesis_in_Arabidopsis.png
- Fig 2: httpscommons.wikimedia.orgwikiFileDsRNA_(1H1K).png
- Figura 3: httpscommons.wikimedia.orgwikiFileRNAi.jpg