Che cos’è il MinION?
Il MinION è uno strumento unico nel suo genere, prodotto e commercializzato dall’azienda Oxford Nanopore Technologies (ONT) a partire dal 2012. Negli ultimi anni questa tecnologia ha raggiunto una forte notorietà grazie alle sue caratteristiche uniche di innovazione, flessibilità e portabilità.
Si tratta di un sequenziatore portatile, di piccole dimensioni, alimentato tramite una semplice porta USB (Fig. 1) e caratterizzato da un’elevatissima processività (sequenziamento high throughput). Questo permette di sequenziare grandi frammenti di DNA (o RNA) in breve tempo, generando reads, o letture, con una lunghezza nell’ordine delle megabasi (Mb). Con una singola analisi è quindi possibile ricostruire la sequenza di frammenti formati da più di 1 milione di nucleotidi.

Il sequenziamento di terza generazione
Il MinION sfrutta una tecnologia di sequenziamento di terza generazione, superando ampiamente la metodologia Sanger e mostrando caratteristiche più interessanti rispetto alle moderne NGS (Next Generation Sequencing) di seconda generazione.
Alla base del MinION vi è il sequenziamento a nanopori. In questa tecnica si utilizzano dei nanopori inseriti in una membrana lipidica o sintetica, dove formano dei canali attraverso cui le molecole possono passare. Ai capi della membrana è applicata una differenza di potenziale che determina la creazione di una corrente elettrica.
Durante il transito all’interno del nanoporo, la molecola blocca il passaggio degli ioni causando una perturbazione di corrente (Fig 2). Questa viene rilevata da un computer ed è diversa per ogni nucleotide. Tramite la lettura del segnale elettrico e delle sue variazioni è possibile identificare la sequenza con cui i diversi nucleotidi sono disposti nella molecola.
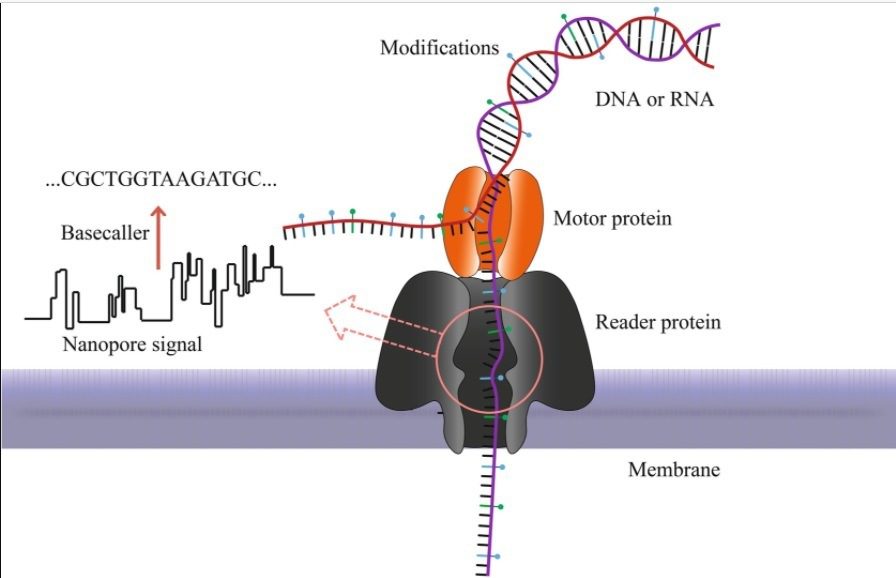
I nanopori sfruttati possono essere di diverso tipo: biologici, allo stato solido o ibridi. Attualmente in Italia vengono commercializzati solo quelli biologici, principalmente di origine batterica. Questi vengono selezionati per poter essere inseriti nella membrana, essere prodotti su larga scala e permettere il passaggio delle macromolecole.
Nel MinION si utilizza l’alfa emolisina (αHL), una tossina emolitica prodotta da Staphylococcus aureus, con un diametro interno di 1 nm (100.000 volte più piccolo di un capello umano). Un’altra proteina batterica utilizzabile è la porina A (MspA), prodotta da Mycobacterium smegmatis, che ha un diametro interno minore rispetto all’αHL.
Preparazione del campione
Come per le altre NGS, è necessario costruire la library di sequenziamento a partire dall’inserto, ovvero la sequenza che si vuole determinare. La libreria può essere di DNA o di RNA, a seconda che si desideri portare avanti un’analisi genomica o trascrittomica.
Il processo di costruzione della libreria è diverso in base alla tecnologia, ma in ogni caso è costituito da 3 step principali:
- La frammentazione del campione ad una lunghezza predefinita;
- L’aggancio degli adapters alle estremità dei frammenti;
- La quantificazione della libreria attraverso le tecniche di PCR;
Un punto a favore della tecnologia MinION è sicuramente la preparazione del campione, che in questo caso è un processo semplice e intuitivo. L’azienda fornisce dei kit ideati appositamente per il processo di sample prep. Questi seguono protocolli differenti, hanno tempi di esecuzione caratteristici e, per questo, incidono diversamente sull’evoluzione del processo. Gli adattatori possono essere aggiunti alla molecola tramite un classico processo di ligazione (60 minuti) oppure usando delle trasposasi specifiche (10 minuti) (Fig 3).

Anche se il protocollo di preparazione della libreria segue gli stessi step chiave, la natura degli adattatori è diversa nelle varie metodologie. Negli adattatori usati in MinION c’è la presenza di due elementi chiave: il tether e la proteina motrice ad attività elicasica. Il primo elemento favorisce l’avvicinamento della libreria al poro, ma la sua presenza è opzionale. L’elicasi, invece, lega fisicamente il DNA al nanoporo e svolge la doppia elica in modo tale da far passare un solo filamento. Inoltre, ha il compito di rallentare il transito dell’acido nucleico all’interno del canale, per permettere una rilevazione efficiente.
Meccanismo di funzionamento e risultati
Leggendo il segnale elettrico è possibile associare ad ogni variazione una singola base azotata. Il processo di associazione è svolto in modo autonomo da un computer che, utilizzando algoritmi specifici definiti basecaller, fornisce direttamente la sequenza letta (Video 1).
Il MinION rappresenta uno dei pochi sequenziatori che permette di effettuare un’analisi dei meccanismi epigenetici. Infatti, la presenza di una metilazione su una base genera un segnale caratteristico, diverso rispetto alla perturbazione associata alla stessa base non metilata.
La rilevazione e l’emissione dei dati avvengono in tempo reale (real time analysis) con una lettura media di 400 basi per secondo. Questo è possibile perché ogni nanoporo analizza le molecole nel campione in maniera indipendente rispetto agli altri. In questo modo si possono sia caricare campioni diversi, ma anche analizzare lo stesso campione più volte per ottenere un accuratezza maggiore.
Vantaggi e limiti del sequenziamento a nanopori
La tecnologia di sequenziamento a nanopori presenta notevoli vantaggi in quanto:
- non bisogna marcare i frammenti;
- produce letture molto lunghe in breve tempo;
- non richiede amplificazione dei frammenti, evitando così tutti i limiti della PCR;
- non necessita di una preparazione complicata del campione né di grandi quantità dello stesso.
Allo stesso tempo, sequenziare tramite questa tecnologia significa dover fare fronte ad un tasso di errore maggiore e alla produzione di un numero di dati inferiore rispetto alle altre NGS, con conseguente aumento dei costi.
Esempi applicativi del sistema MinION
Tramite il MinION è possibile: sequenziare interi genomi o regioni target (DNA barcoding), sequenziare l’RNA, portare avanti studi metagenomici identificando nuove specie e studiare i meccanismi epigenetici (metilazione o RNA non coding).
La flessibilità, la versatilità e soprattutto la portabilità del MinION si riflettono in una crescita costante della letteratura degli ultimi anni. Solo nei primi mesi del 2021, ad esempio, il MinION è stato utilizzato per: caratterizzare il microbiota intestinale umano, sequenziare il genoma del SARS-CoV-2 direttamente da tamponi orofaringei e identificare nuovi microrganismi da campioni marini.
Dall’Antartide allo spazio
Il sequenziatore portatile è inoltre capace di resistere a condizioni ambientali estreme ed è per questo utilizzabile direttamente sul campo, evitando il trasporto dei campioni e riducendo notevolmente il tempo di analisi. Grazie al MinION, infatti, si sono costruiti laboratori da campo nelle foreste tropicali di Tanzania, Ecuador e Brasile, nella savana africana e nelle distese ghiacciate dell’Antartide.
Nel 2016 il MinION è addirittura arrivato nello spazio, a bordo dello Stazione Spaziale Internazionale (ISS), con lo scopo di monitorare la salute dell’equipaggio (Fig 4). In seguito ad un viaggio spaziale, infatti, gli astronauti mostrano spesso una risposta immunitaria disregolata e una maggior suscettibilità ai patogeni. In questo contesto, il sequenziatore ha permetto di effettuare diagnosi di infezioni virali ma anche valutare i livelli di espressione genica e l’accumulo di mutazioni.

I capi applicativi del sequenziamento a nanopori sono quindi moltissimi, ma tanti altri sono quelli non ancora esplorati. Come l’azienda stessa afferma «our goal is to enable the analysis of any living thing, by anyone, anywhere». Possiamo sicuramente affermare di non essere molto lontani da questa visione.
Fonti
- Oxford Nanopore Technologies
- A Rapid and Accurate MinION-Based Workflow for Tracking Species Biodiversity in the Field (mdpi.com)
- Applications of Oxford Nanopore Sequencing in Schizosaccharomyces pombe
- Full-length 16S rRNA gene amplicon analysis of human gut microbiota using MinION™ nanopore sequencing confers species-level resolution
- Microbial diversity characterization of seawater in a pilot study using Oxford Nanopore Technologies long-read sequencing | BMC Research Notes
- Nanopore DNA Sequencing and Genome Assembly on the International Space Station