In un organismo, l’apoptosi o morte programmata di una cellula è un processo finemente controllato che porta all’eliminazione della cellula senza arrecare danni alle cellule circostanti. Le cellule in apoptosi presentano diversi cambiamenti a livello del nucleo tra cui: degradazione dell’involucro nucleare, blebbing nucleare, condensazione della cromatina e frammentazione del DNA.
Durante l’apoptosi, il DNA è degradato da endonucleasi in frammenti lunghi 180-200 bp. Il saggio TUNEL (Terminal deoxynucleotidyl transferase dUTP Nick End Labeling) marca i punti di rottura del doppio filamento di DNA (Double Strand Breaks, DSBs). Attualmente, è un saggio largamente utilizzato per rilevare cellule apoptotiche.
Marcare il DNA frammentato
Durante le fasi finali dell’apoptosi si ha la frammentazione del DNA. Le endonucleasi creano sul DNA estremità 3′-OH che possono essere substrato dell’enzima terminal deoxynucleotidyl transferase (TdT) per attaccare dUTP modificati tra cui EdUTP e BrdUTP. Quindi il DNA frammentato viene marcato con nucleotidi modificati che possono essere riconosciuti con differenti strategie.

Metodo
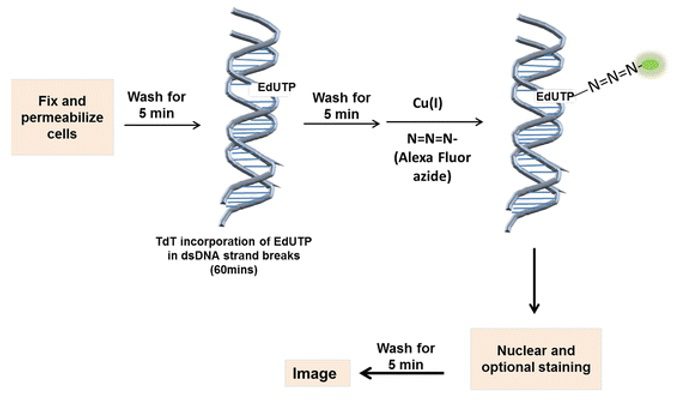
Le cellule o il tessuto da analizzare sono fissati e permeabilizzati per preservare le cellule apoptotiche nello stadio finale del processo. In particolare, la fissazione serve ad evitare la possibilità di falsi negativi (assenza di apoptosi) dovuti a distaccamento e perdita di cellule del campione in esame.
Al momento, nel saggio TUNEL viene impiegato il nucleotide modificato EdUTP (5-Ethynyl-dUTP) che contiene un gruppo alchino (triplo legame tra due carboni). Come spiegato sopra, EdUTP è incorporato all’estremità 3′-OH da parte dell’enzima TdT. Successivamente, al campione viene aggiunto una molecola fluorescente contenente un gruppo azide (N=N=N-) come Alexa Fluor Azide. Il nucleotide modificato e l’azide fluorescente reagiscono tra loro in una click-reaction (reazione a scatto) di cicloaddizione azide-alchino catalizzata dal rame. Pertanto possiamo evidenziare tutti i frammenti di DNA che hanno incorporato il nucleotide modificato. Questa reazione è altamente specifica, perchè i gruppi che reagiscono (azide e alchino) non sono presenti in ambiente biologico, in molecole biologiche, in cellule, tessuti o organismi modello.
La molecola fluorescente coniugata al nucleotide può essere utilizzata per la rilevazione diretta del DNA frammentato attraverso metodi basati sulla fluorescenza: microscopia a fluorescenza o citometria a flusso.
In breve, il saggio TUNEL prevede i seguenti passaggi:
- Fissazione e permeabilizzazione del campione;
- Incorporazione di EdUTP da parte dell’enzima TdT;.
- Click-reaction tra EdUTP e azide fluorescente;
- Rilevazione del segnale fluorescente.
Questo metodo risulta nella rilevazione di una percentuale maggiore di cellule apoptotiche in meno di due ore!
Risultati attesi
Il saggio TUNEL viene applicato al campione controllo e al campione test differenti per una condizione o un trattamento che si ipotizza influenzi l’apoptosi cellulare. Confrontando la fluorescenza dei diversi campioni si può confermare se l’apoptosi aumenta o diminuisce in seguito ad un trattamento specifico.
Limitazioni del saggio TUNEL
La frammentazione del DNA è una caratteristica comune a necrosi e apoptosi, quindi è importante utilizzare un secondo metodo di analisi per confermare l’apoptosi. Questo secondo metodo deve essere indipendente dal saggio TUNEL, cioè non valutare la frammentazione del DNA, ma altre caratteristiche dell’apoptosi. Ad esempio, possono essere utili:
- immunoistochimica per individuare la Caspasi 3;
- western blot contro PARP (Poli ADP-ribosio polimerasi) tagliate dalle caspasi;
- colorazione con Annexin V per rilevare fosfatidil-serina sulla superficie cellulare.
Fonti
- https://www.thermofisher.com/it/en/home/life-science/cell-analysis/cellular-imaging/fluorescence-microscopy-and-immunofluorescence-if/imaging-apoptosis-assays/tunel-assays-dna-fragmentation-chromatin-condensation.html
- https://www.creative-bioarray.com/support/tunel-apoptosis-assay-fluorescent.htm
- https://link.springer.com/protocol/10.1007/978-1-61779-860-3_5
- https://www.clinicacastelli.it/enciclopedia/apoptosi/
Crediti immagini
- Immagine in evidenza: https://dietapaleo.it/blog/che-cose-l-apoptosi/
- Figura 1: https://www.jenabioscience.com/probes-epigenetics/dna-cdna-labeling/click-chemistry-based-dna-cdna-labeling/click-functionalized-nucleotides/clk-t07-5-ethynyl-dutp-5-edutp
- Figura 2: https://www.creative-bioarray.com/support/tunel-apoptosis-assay-fluorescent.htm