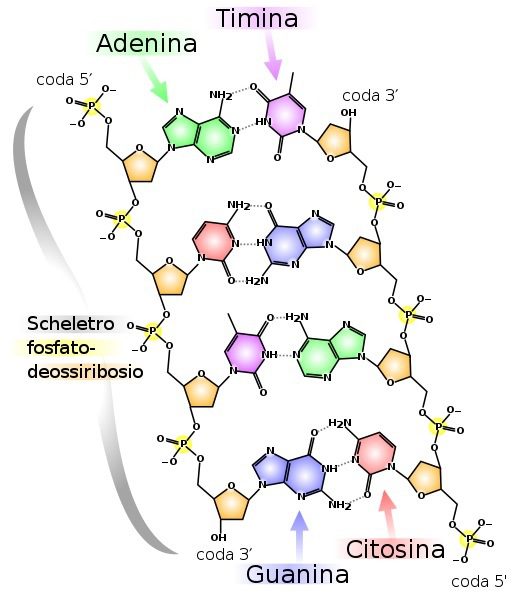
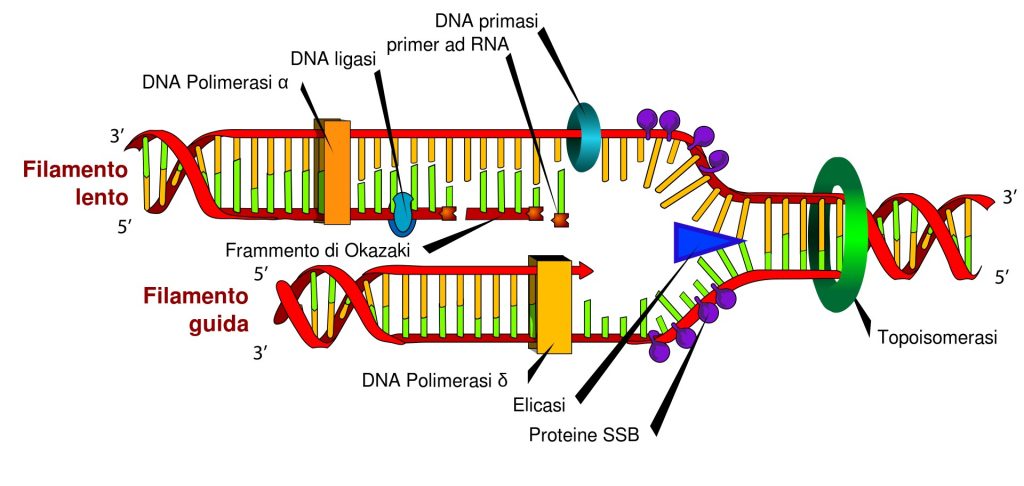
Le mutazioni nel DNA (fig. 1) sono frequenti, e sono causate da errori durante la replicazione della doppia elica (fig. 2) o a causa di sostanze mutagene. Consistono nell’errato appaiamento di basi nucleotidiche o problemi nella replicazione, e sono altamente instabili. Possono infatti essere tramandate alle generazioni successive, causando a volte importanti e numerosi danni. Per questo motivo, la cellula ha sviluppato dei meccanismi di riparazione dei danni al DNA, in modo da impedirne le alterazioni che possono avere un impatto anche deleterio per l’organismo.
I meccanismi di riparazione dei danni al DNA possono essere suddivisi in due principali categorie: i sistemi di riparazione per correzione diretta, e i sistemi di riparazione per escissione.
La differenza principale tra questi due macrogruppi è che nel caso del primo, sistemi enzimatici della cellula riparano le regioni danneggiate ripristinando il DNA al suo stato precedente, mentre, nel caso della riparazione per escissione, la regione danneggiata viene rimossa e questo gap viene colmato da nuovo DNA risintetizzato.
Meccanismi di correzione diretta
Esistono diversi meccanismi che la cellula mette in atto, sia durante la duplicazione del DNA che appena successivamente, per riparare danni che avvengono per un errato appaiamento di nucleotidi ad opera della DNA Polimerasi. Quest’enzima infatti, compie errori nell’inserimento dei nucleotidi con una frequenza di 10-5 errori per generazione.
Riparazione diretta ad opera della DNA Polimerasi
L’enzima DNA Polimerasi, fondamentale nella duplicazione del genoma, ha anche un’attività di correzione di bozze. Se viene inserito un nucleotide scorretto, questa ritorna indietro per correggere l’errore e appaiare la giusta base azotata, per poi riprendere l’attività di duplicazione nella giusta direzione di sintesi.
Riparazione dei dimeri di pirimidina indotti da UV
I raggi ultravioletti possono causare l’insorgenza di dimeri di timina o di altre basi pirimidiniche. Un dimero di timina si forma a causa di un legame covalente tra atomi di carbonio C5 e C6 di due timine adiacenti. Questa reazione di formazione del legame covalente è spesso catalizzata dalle radiazioni UV aventi lunghezza d’onda compresa tra 260 nm e 280 nm, o dalla presenza di agenti mutageni. Il meccanismo di riparazione diretta prevede la fotoriattivazione (fig. 3) un meccanismo in cui un enzima chiamato fotoliasi scinde il dimero di timina attivato da un fotone di luce, a seguito dell’esposizione alle lunghezze d’onda tra i 320 e 370 nm. La fotoliasi non è un enzima rinvenuto nell’uomo, ma è stato caratterizzato in procarioti e eucarioti inferiori.
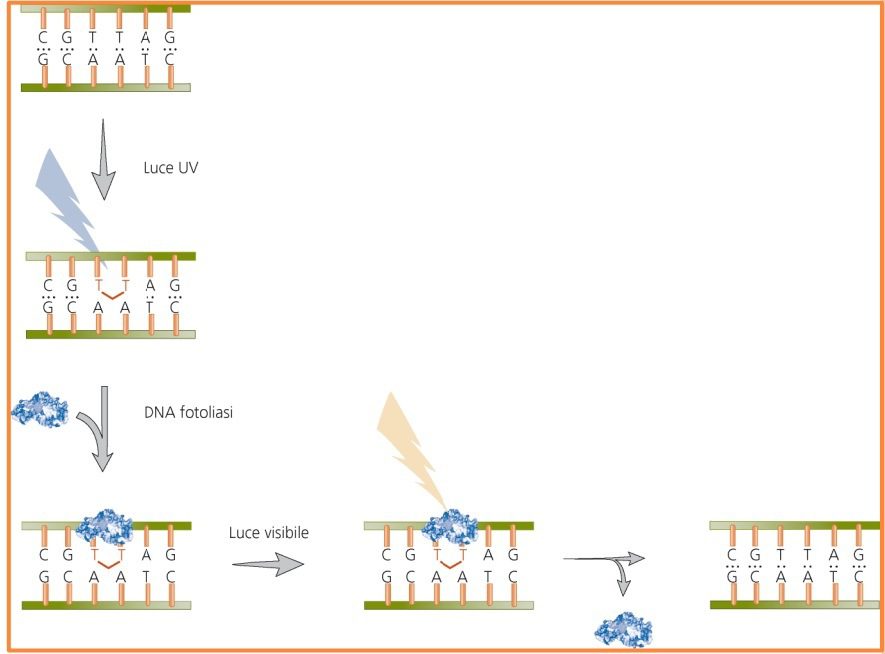
Riparazione dei danni da alchilazione:
Come identificato in E. Coli, un enzima chiamato O6-metilguanina metiltranferasi si occupa di rimuovere il gruppo metilico dalla guanina a seguito di un’alchilazione di questa, che può verificarsi a causa del trasferimento di gruppi alchilici alle basi. Diversi farmaci oncologici sono agenti alchilanti.
Meccanismi di riparazione per escissione
Quando il danno al DNA interessa un solo dei due filamenti dell’elica, quello non danneggiato può essere utilizzato come stampo, mentre l’altro viene “corretto” nel punto del danno. Ciò che accade è una rimozione, o di basi o di nucleotidi errati, e una nuova sintesi e inserimento della porzione corretta.
Riparazione per escissione di basi (BER)
Se una base è stata danneggiata, interviene l’enzima glicosilasi che riconosce dapprima l’errore, e poi rimuove la base tagliando il legame tra la base e lo zucchero. Una volta che anche altri enzimi saranno interceduti per disgregare anche il legame zucchero-fosfato, nella catena del DNA ci sarà una discontinuità. Questa sarà colmata dall’inserimento di una nuova base, stavolta corretta, dalla DNA Polimerasi, e il legame saldato grazie alla DNA Ligasi.
Riparazione per escissione di nucleotidi (NER)
Così come le basi azotate possono essere rimosse, anche l’intero nucleotide può essere sostituito. Se, ad esempio, a causa dei raggi UV si formano dei dimeri di timina ma non è possibile la fotoriattivazione, uno o più nucleotidi possono essere escissi e sostituiti. Questo avviene ad opera di una serie di fattori proteici. Le fasi sono simili alla riparazione per escissione di basi. Esistono due diverse modalità di intervento: la riparazione accoppiata alla trascrizione (Transcription Coupled Repair, TCR), se il danno viene riconosciuto dalla RNA Polimerasi durante la trascrizione, e riparazione del genoma globale (Global Genome Repair, GGR), in cui diversi enzimi agiscono in sinergia per riconoscere il danno al DNA, rimuovere i tratti danneggiati e sostituirli.
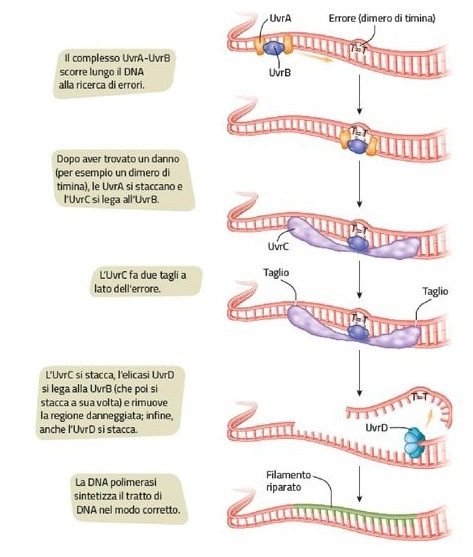
In E. Coli le proteine coinvolte in questo processo sono e UvrA, UvrB, UvrC e UvrD (Fig. 4).
Il sistema mismatch repair (MMR)
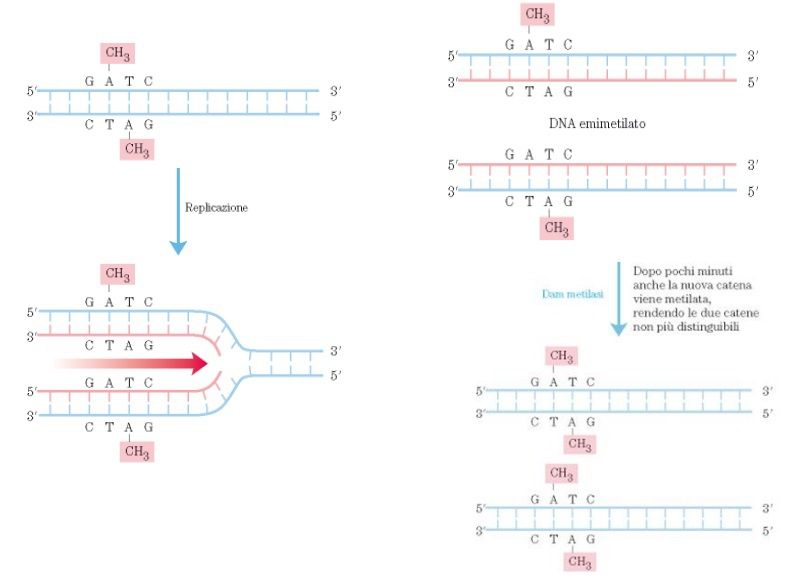
In questo processo, è il nucleotide appaiato erroneamente che viene sostituito. Affinché avvenga questo, il sistema di correzione degli errori di appaiamento (ad esempio una timina appaiata con una guanina e non con un’adenina) deve essere in grado di riconoscere in quale dei due filamenti è presente l’errore. L’errore viene identificato dallo stato di metilazione di una sequenza GATC in prossimità. Se la sequenza non è metilata, la porzione di DNA contenente la base errata viene escisso e sostituito da nuovo DNA (fig. 5).
Patologie legate a difetti nella replicazione e riparazione del DNA
Difetti nei meccanismi di riparazione del DNA sono responsabili di alcune malattie nell’uomo. Alcuni esempi sono lo xeroderma pigmentosum, una patologia che causa l’insorgenza di tumori maligni della pelle ed elevata fotosensibilità, e l’atassia telangectasia, una malattia che colpisce da 1 su 40.000 fino a 1 su 100.000 nati vivi, caratterizzata da immunodeficienza, difetti nella coordinazione muscolare e progressiva atrofia muscolare spinale. Altre patologie associate a difetti nei meccanismi di riparazione della doppia elica sono l’anemia di Fanconi, la sindrome di Bloom e la sindrome di Cockayne. Anche il cancro al colon non-poliposico ereditario (HPNCC) si sviluppa quando insorgono mutazioni nei geni coinvolti nel meccanismo di mismatch repair.
Fonti:
- Genetica. Un approccio molecolare. Peter J. Russell
- Nelson & Cox – I principi di Biochimica di Lehninger- Zanichelli
- https://www.news-medical.net/life-sciences/Mechanisms-of-DNA-Repair-(Italian).aspx