In collaborazione con: Claudio Bandi, PhD, Professore Ordinario di Microbiologia, Dipartimento di Bioscienze e CRC Pediatrico Romeo ed Enrica Invernizzi, Università degli Studi di Milano.
Diagnosi microbiologica attuale e sequenziamento del DNA
I reparti di terapia intensiva si trovano spesso a combattere infezioni da batteri resistenti agli antibiotici. Tra questi ricordiamo Pseudomonas aeruginosa, Eschericha coli, Klebsiella pneumoniae, Staphylococcus aureus. Inoltre, rivestono una certa importanza anche le infezioni da parte di microrganismi classificabili come funghi, appartenenti al genere Candida. La diagnosi e, quindi, la scelta del trattamento durante un processo infettivo sostenuto da microrganismi resistenti a determinati antibiotici, è spesso una lotta contro il tempo. Le moderne tecniche di sequenziamento massivo del DNA offrono per la prima volta la possibilità di introdurre la genomica nella microbiologia clinica. Il sequenziamento e l’analisi del DNA hanno acquisito oggi una velocità e una robustezza tale da proporli come valide alternative all’identificazione microbiologica classica.
Metodi classici di identificazione
Alcuni dei metodi di identificazione microbiologica più usati attualmente si basano su:
- coltura in vitro dell’organismo su terreni specifici;
- antibiogrammi, che danno un’informazione preziosa nella scelta del trattamento antibiotico;
- identificazione della specie microbica con una metodologia rapida nota come MALDI-TOF;
- reazione a catena della polimerasi (dall’inglese: Polymerase Chain Reaction, PCR);
- osservazione microscopica.
Inoltre, per la diagnosi di infezioni da parte di microrganismi non facilmente coltivabili o difficili da isolare, per una osservazione diretta viene utilizzata una metodologia che rileva la risposta del nostro organismo nei confronti di determinate specie microbiche. Si tratta dell’ELISA (dall’inglese: Enzyme-Linked Immunosorbent Assay);
L’obiettivo principale della diagnosi precoce è sicuramente la riduzione dei tempi di trattamento antibiotico e l’aumento della sua efficacia e specificità. Solo recentemente le tecniche di diagnosi basate sul sequenziamento del DNA sono entrate a far parte delle procedure per la diagnosi microbiologica nei laboratori più attrezzati.
Limiti dei metodi di identificazione
Gli agenti patogeni emergenti o riemergenti, provenienti per esempio da un processo di spill-over, o che non sono “attesi” in un determinato contesto epidemiologico, possono sfuggire all’identificazione mediante i metodi sopra elencati. Anche i frequenti processi di acquisizione di fattori di resistenza agli antibiotici, per trasferimento orizzontale da un organismo a un altro, possono complicare la diagnosi, producendo falsi negativi o situazioni in cui il microrganismo risulta non identificabile. Inoltre, i metodi tradizionali per la diagnosi microbiologica non permettono di seguire l’evoluzione epidemiologica dell’agente patogeno. Da un un punto di vista strategico, infatti, risulta di grande interesse per la microbiologia clinica seguire l’evoluzione e l’espansione di eventuali varianti genomiche.
Nella maggior parte dei casi, l’identificazione microbiologica è una pratica routinaria. Spesso la scelta dell’antibiotico più adatto a trattare l’infezione si basa sul profilo di resistenza ottenuto mediante un antibiogramma. In generale, i metodi basati sulla coltura dei microrganismi possono ritardare la diagnosi, specialmente nel caso di organismi a crescita lenta, ad esempio i micobatteri responsabili della tubercolosi. Quanto più veloce sarà la scelta corretta della terapia antibiotica, tanto più rapidamente si bloccherà l’infezione evitando eventuali coinfezioni o la selezione di ceppi resistenti.
Genomica in microbiologia clinica mediante NGS
I metodi di sequenziamento del DNA di ultima generazione, conosciuti come Next Generation Sequencing (NGS), permettono identificare gli organismi usando il loro genoma. Leggendo il genoma, questi metodi svelano la presenza di fattori di resistenza già conosciuti insieme ad altri di nuova scoperta o che non ci si aspetterebbe trovare. I moderni flussi di analisi bioinformatica si alimentano in tempo reale dai dati provenienti dei sequenziatori di DNA. In questo modo la genomica applicata alla microbiologia clinica, permette comparare in streaming le sequenze prodotte con banche dati specializzate alla ricerca di fattori di resistenza, virulenza o ottenendo informazioni sull’identificazione precisa dell’organismo in esame.
Questi metodi offrono la possibilità di diagnosticare la presenza di un determinato patogeno senza neppure aspettare la sua crescita in coltura. Ad esempio, un approccio noto come metagenomica, basato sul sequenziamento massivo e casuale di frammenti di DNA, permette di campionare frammenti genomici, sequenziarli e annotarli praticamente in tempo reale. Durante le epidemie di Ebola o Zika questi metodi hanno permesso identificare l’agente patogeno a una velocità e precisione mai vista prima.
Sequenziamento e diagnosi in tempo reale, Ebola e Zika
Uno dei metodi di sequenziamento di terza generazione conosciuto come MinION, dell’azienda Oxford Nanopore Technologies, ha inoltre il grande vantaggio della portabilità. Questo sequenziatore, delle dimensioni di un pendrive, si controlla direttamente da un computer portatile collegandolo a una entrata USB (Universal Serial Bus 3.0). Questo sequenziatore si può acquistare facilmente a un un costo iniziale inferiore ai 1.000€. MinION permette un sequenziamento sul campo senza la necessità di un laboratorio di appoggio sofisticato. Anche l’analisi bioinformatica associata può essere facilmente condotta sullo stesso computer portatile, limitando l’unica necessità a una fonte di energia (Figura 1).

Questo tipo di configurazione ha permesso l’analisi in tempo reale del virus Ebola durante l’epidemia del 2014-2016 in Liberia e Guinea. Il sequenziatore MinION si trova inoltre alla base del progetto ZiBRA-Project che segue l’evoluzione del virus Zika in Brasile dal 2016.
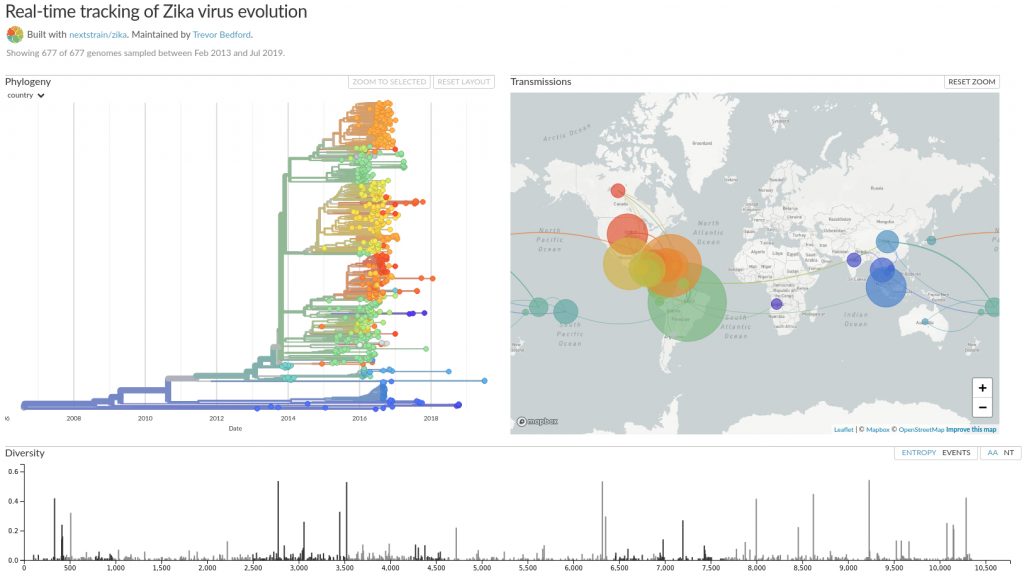
I dati genomici ottenuti dal sequenziamento dei campioni clinici hanno permesso stabilire l’evoluzione filogenetica, spaziale e temporale durante le epidemie in tempo quasi reale. Una delle piattaforme di grande successo per l’analisi di questi dati genomici è sicuramente Nextstrain. Questo progetto open-source, nasce per sfruttare il potenziale scientifico dei dati genomici di patogeni sequenziati nel campo della sanità pubblica. Nexstrain, partendo dai genomi virali sequenziati, permette migliorare la risposta sanitaria alle epidemie mediante varie applicazioni di visualizzazione filogenetica, genomica, spaziale e temporale (Figura 2). Questo tipo di strumenti sarà indubbiamente essenziale per l’introduzione della genomica nella microbiologia clinica.
Possibilità di introduzione dei metodi NGS
Il sequenziamento mediante NGS di terza generazione informa sull’agente patogeno non appena un frammento del suo DNA (o cDNA nel caso di virus a RNA) viene letto e trasformato in un file. Allo stesso tempo in cui il dato viene prodotto, questo si incanala nelle pipelines di analisi attivando una ricerca istantanea nelle banche dati specifiche. Un eventuale allineamento con un patogeno, fattore di resistenza o virulenza, farà quindi scattare un campanello d’allarme dichiarando la sua presenza.
Sebbene questi metodi siano molto promettenti, l’uso della genomica per l’identificazione microbiologica in microbiologia clinica è ancora oggi in fase iniziale. I costi di sequenziamento per campione si possono considerare ancora alti comparati con i metodi tradizionali. È comunque importante considerare che la quantità e qualità dei dati aumenta continuamente, riducendo i costi per campione. Anche l’automatizzazione dell’analisi bioinformatica gioca a favore dell’introduzione del sequenziamento nella diagnosi clinica microbiologica, garantendo risultati ripetibili e statisticamente robusti.
A livello locale, l’uso delle NGS nei laboratori di microbiologia dei centri sanitari può rappresentare un aiuto fondamentale all’identificazione. A livello globale invece, una moderna epidemiologia molecolare basata sull’uso delle NGS permetterà ottenere dati sull’espansione ed evoluzione del patogeno in tempo quasi-reale. Una microbiologia clinica basata sull’uso delle NGS permetterà, inoltre, di disegnare mappe spaziali e temporali dell’espansione di agenti infettivi, basandosi sull’evoluzione del loro genoma completo. In questo modo sarà possibile informare tempestivamente le agenzie sanitarie competenti sui rischi di nuove varianti o sulla comparsa di fattori di resistenza o virulenza. Questi dati saranno di fondamentale importanza nell’elaborazione dei piani strategici che potrebbero cambiare l’esito delle epidemie e pandemie presenti o future.
Sull’autore: Dr. Giuseppe D’Auria, PhD. (ORCID, gidauria), responsabile del Servizio di Bioinformatica della fondazione FISABIO, Valenzia, Spagna. Progetto “Microonstream: Life under the microscope” (YT, Twitter).
Fonti
- D’Auria G, Schneider MV, Moya A. Live genomics for pathogen monitoring in public health. Pathogens. 2014 Jan 21;3(1):93-108.
- Gardy JL, Loman NJ. Towards a genomics-informed, real-time, global pathogen surveillance system. Nat Rev Genet. 2018 Jan;19(1):9-20.
- Hoenen T, Groseth A, Rosenke K, et al. Nanopore Sequencing as a Rapidly Deployable Ebola Outbreak Tool. Emerg Infect Dis. 2016 Feb;22(2):331-4.
- Quick J, Loman NJ, Duraffour S, et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 2016 Feb 11;530(7589):228-232.
- Nextstrain, Genomic epidemiology of the 2013-2016 West African Ebola epidemic: https://nextstrain.org/ebola.
- Nextstrain, Real-time tracking of Zika virus evolution: https://nextstrain.org/zika.
- ZiBRA project: real-time sequencing of Zika virus in Brazil: www.zibraproject.org.